Studium polymorfismu u vybraných populací smrku ztepilého Picea abies (L.)
|
|
|
- Vojtěch Dvořák
- před 8 lety
- Počet zobrazení:
Transkript
1 Výzkum možností minimalizace obsahů organických škodlivin ve zdrojích pitných vod v Krušných horách Studium polymorfismu u vybraných populací smrku ztepilého Picea abies (L.) Karsten pomocí DNA analýz Ing. Helena Cvrčková, Ph.D., Ing. Pavlína Máchová, Ph.D. Září 2013 Výzkumný ústav lesního hospodářství a myslivosti, v.v.i., cvrckova@vulhm.cz, machova@vulhm.cz
2 Autor: Ing. Helena Cvrčková, Ph.D., Ing. Pavlína Máchová, Ph.D. Výzkumný ústav lesního hospodářství a myslivosti, v.v.i. Strnady 136, Jíloviště, Doručovací pošta: Praha 5 Zbraslav cvrckova@vulhm.cz, machova@vulhm.cz Foto: M. Krtička
3 Obsah Úvod 3 Materiál a metodika 4 Protokol izolace 6 RAPD 6 PCR - RFLP 7 SSR markery 9 Výsledky 11 Diskuze 18 Závěr 19 Literatura 20 2
4 Úvod Smrk ztepilý Picea abies (L.) Karsten je z ekonomického i ekologického hlediska jednou z nejvýznamnějších dřevin, do Evropy migroval pravděpodobně z východní Asie a v posledních dvou stoletích byl intenzivně vysazován v centrální Evropě (SCHUBERT et al. 2001). Na našem území jsou těžištěm původního rozšíření okrajová horstva. Protože má povrchovou kořenovou soustavu, je značně náročný na půdní vlhkost a vyžaduje i vyšší relativní vlhkost vzduchu. Na půdu a geologické podloží nemá velké nároky. Nedostatek vláhy je limitujícím faktorem jeho dobrého růstu. Snese i nadbytečnou vlhkost rašelinišť (ÚRADNÍ- ČEK et al. 2009). Ale na chudých křemitých půdách a kyselých rašelinách roste špatně, protože těžko snáší nedostatečně provzdušněné půdy (CHMELAŘ 1980). Stabilita lesních porostů ovlivňuje možnost plnění jejich vodohospodářské a půdoochranné funkce a stabilní porost může regulovat případné vyplavování organických látek. Cílem práce bylo zjistit na základě úrovně polymorfismu sledovaných porostů z oblasti Krušných hor (Fláje a povodí potoka Rauschenbachu Sasko) jejich stabilitu, porovnáním míry jejich variability zhodnotit schopnost dřevin přizpůsobit se extrémním klimatickým podmínkám lokality a vyššímu obsahu rašeliny. Bylo vybráno 5 porostů z oblasti Fláje a dva porosty z povodí potoka Rauschenbachu, porosty se vyskytují na rašelinných nebo kamenitých půdách, viz. tab. 1. Pro možnost porovnání genetických charakteristik porostů z této oblasti Krušných hor byly do studie zahrnuty i dvě původní populace chlumního ekotypu smrku ztepilého a to z oblasti Českosaského Švýcarska a Křivoklátska a dále jedna populace horského ekotypu z Orlických hor z oblasti Trčkova. Vývoj přirozené populace souvisí s její genetickou diverzitou, čím jsou populace v genetické výbavě rozmanitější, tím se lépe přizpůsobí měnícím se životním podmínkám. V naší studii bylo také testováno zda různé úrovně vitality vybraných porostů jsou podmíněny i mírou genetické variability, tab. 1. Genetické vlastnosti vybraných populací byly studovány pomocí analýz DNA. DNA markery umožňují přímo studovat genom organismů, jsou založeny na polymorfismu sekvencí nukleotidů a nejsou ovlivněny podmínkami prostředí jako například izoenzymové markery. Významným přínosem pro rozvoj analýz DNA byl objev polymerázové řetězcové reakce, o který se zasloužil Kary Mullis v roce 1985 (BROWN 2006). Při této reakci se mnohonásobně namnoží malý úsek DNA, který lze následně detekovat a studovat. Většina současných metod studia DNA markerů je založena na PCR polymerázové řetězové reakci, při které dochází k selektivní amplifikaci určitého úseku nukleové kyseliny. PCR je třístupňový proces v několika opakujících se cyklech, při kterém termostabilní DNA polymeráza namnoží úsek mezi dvěma oligonukleotidy (primery) na základě komplementarity bází se sekvencí templátové DNA. Při PCR reakci je využívána schopnost DNA opakovaně denaturovat a reasociovat v závislosti na různé teplotě. Směs, v níž PCR probíhá, obsahuje templátovou DNA, většinou dva různé primery, nukleotidtrifosfáty, termostabilní polymerázu a reakční pufr. Vzájemné poměry a koncentrace templátu, primerů a především hořečnatých iontů jsou v průběhu PCR klíčové. Velmi často je nezbytné optimalizovat složení reakční směsi tak, aby výtěžky namnožených úseků DNA byly dostatečně vysoké a spolehlivé. PCR reakce se provádí pomocí teplotních cyklovačů, které automaticky opakují nastavené teplotní cykly a za krátkou dobu se vytvoří miliardy kopií daného úseku DNA. Je to rychlá a specifická metoda, která má široké uplatnění při molekulárně - genetických studiích v mnoha oborech. Pro získání informací o genetické proměnlivosti zkoumaných vzorků je potřebné vyhledat DNA markery vykazující polymorfis- 3
5 mus. Míry charakterizující genetickou strukturu a proměnlivost populací jsou založeny na alelických frekvencích jednotlivých alelických variant v lokusech (PAULE 1992). Pro testování polymorfismu vybraných populací smrku ztepilého byly využity DNA markerovací systémy (metody) RAPD (Random Amplified Polymorphic DNA), PCR RFLP (Restriction Fragment Lenght Polymorphis) pro mitochondriální úseky DNA a mikrosatelity - SSR (simple sequence repeats). Polymorfismus byl pozorován především u několika mikrosatelitových lokusů. K těmto polymorfním mikrosatelitovým lokusům se nechaly fluorescenčně označit primery a u amplifikačních produktů byla pro zjištění jejich přesných velikostí provedena fragmentační analýza na genetickém analyzátoru. Pomocí mikrosatelitů, které patří ke kodominantním markerům lze zjišťovat heterozygotnost, jednu ze základních charakteristik genetické proměnlivosti. Heterozygotnost vyjadřuje podíl heterozygotních jedinců v daném lokusu z celkového počtu pozorovaných jedinců (PAULE 1992). V rámci studie byla dále statisticky porovnávána genetická podobnost mezi populacemi na základě Neiových genetických vzdáleností, frekvence alel, diferenciace mezi populacemi a byl hodnocen polymorfismus sledovaných populací. Genetická proměnlivost byla testována u vzorků smrku ztepilého z Krušných hor z oblasti Fláje z porostů označených SM1, SM2, SM3, SMA, SMB a u dvou porostů z povodí potoka Rauschenbachu na území Saska označených SMK a SMR. Pro porovnání genetických charakteristik byly testovány i populace chlumního ekotypu smrku ztepilého rostoucího v lokalitách Českosaského Švýcarska a Křivoklátska a vzorky populace smrku horského ekotypu z oblasti Trčkova Šerlišský kotel v Orlických horách. Odběry vzorků byly provedeny u lokalit povodí Rašeliníku (obr. 1; dále jen Fláje) a Povodí potoka Rauschenbach z porostů nacházejících se na rozdílných stanovištích, a to z kamenitých lokalit nebo z lokalit s vyšším obsahem rašeliny. Odběry byly provedeny pro porovnání úrovně polymorfismu s vitálními porosty i z porostu SMA se sníženou vitalitou stromů - prosychající koruny (obr. 2, tab. 1). Odběry výchozího materiálu byly prováděny během roků 2011 a 2012, z Flájí, Saska i Orlických hor byly pro analýzy DNA odebírány jehlice. V případě cenných původních populací chlumního ekotypu smrku ztepilého rostoucích v lokalitách Českosaského Švýcarska a Křivoklátska byla DNA izolována z odvozených embryogenních linií. Embryogenní kultury smrku ztepilého byly založeny ze zygotických embryí nezralých semen a jsou kultivovány v bance explantátů jako zdroj reprodukčního materiálu (MALÁ et al.1995). Materiál a metodika 4
6 Tab. 1: Přehled míst odběrů a označení vzorků smrku ztepilého Lokality odběru smrku ztepilého Označení populace/porostu Označení vzorků Fláje - u bloků (souřadnice N 50,663250, E 13, ) lokalita s vyšším obsahem rašeliny Fláje silnice K Jiříku (souřadnice N 50,657933, E 13, ) kamenitá lokalita Fláje u brány (souřadnice N 50,654433, E 13,5710 ) lokalita s vyšším obsahem rašeliny Fláje oblast A, porost se sníženou vitalitou* (souřadnice N 50,664322, E 13, ) lokalita s vyšším obsahem rašeliny Fláje oblast B, (souřadnice N 50,665717, E 13, ) lokalita s vyšším obsahem rašeliny Povodí Rauschenbachu - Sasko (souřadnice N 50,710633, E 13,58630 ) kamenitá lokalita SM1 SM /1/ 1, SM /1/ 2, SM /1/ 3, SM /1/ 4, SM /1/ 5, SM /1/ 6, SM /1/ 7, SM /1/ 8, SM /1/ 9, SM /1/ 10, SM /1/ 11, SM /1/ 12, SM /1/ 13, SM /1/ 14, SM /1/ 15, SM /1/ 16, SM /1/ 17, SM /1/ 18, SM /1/ 19, SM /1/ 20, SM /1/ 21 SM2 SM /2/ 1A, SM /2/ 2A, SM /2/ 3A, SM /2/ 4A, SM /2/ 5A, SM /2/ 6A, SM /2/ 7A, SM /2/ 8A, SM /2/ 9A, SM /2/ 10A, SM /2/ 11A, SM /2/ 12A, SM /2/ 13A, SM /2/ 14A, SM /2/ 15A,SM /2/ 16A, SM /2/ 17A, SM /2/ 18A, SM /2/ 19A, SM /2/ 20A SM3 SM /3/ 21A, SM /3/ 22A, SM /3/ 23A, SM /3/ 24A, SM /3/ 25A, SM /3/ 26A, SM /3/ 27A, SM /3/ 28A, SM /3/ 29A, SM /3/ 30A, SM /3/ 31A, SM /3/ 32A, SM /3/ 33A, SM /3/ 34A, SM /3/ 35A, SM /3/ 36A, SM /3/ 37A, SM /3/ 38A, SM /3/ 39A, SM /3/ 40A SMA SMA-1, SMA-2, SMA-3, SMA-4, SMA-5, SMA- 6, SMA-7, SMA-8, SMA-9, SMA-10, SMA-11, SMA-12, SMA-13, SMA-14, SMA-15, SMA-16 SMA-17, SMA-18, SMA-19, SMA-20 SMA-21, SMA-22, SMA-23, SMA-24, SMA-25, SMA-26, SMA-27, SMA-28, SMA-29, SMA-30 SMB SMB-1, SMB-2, SMB-3, SMB-4, SMB-5, SMB-6, SMB-7, SMB-8, SMB-9, SMB-10, SMB-11, SMB-12, SMB-13, SMB-14, SMB-15, SMB-16, SMB-17, SMB-18, SMB-19, SMB-20, SMB-21, SMB-22, SMB-23, SMB-24, SMB-25, SMB-26, SMB-27, SMB-28, SMB-29 SMK SMK-1, SMK-3, SMK-5, SMK-7, SMK-9, SMK-11, SMK-13, SMK-15, SMK-17, SMK-19, SMK-21, SMK-23, SMK-25 Povodí Rauschenbachu - Sasko (souřadnice N 50,714483, E 13, ) lokalita s vyšším obsahem rašeliny SMR SMR-1, SMR-3, SMR-5, SMR-7, SMR-9, SMR-11, SMR-13, SMR-15, SMR-17, SMR-19, SMR-21, SMR-23, SMR-25 Českosaské Švýcarsko (souřadnice N 50,886248, E 14, ) Křivoklátsko (souřadnice N 49,988259, E 13, ) Trčkov Šerlišský kotel (Orlické hory), (souřadnice N 50,312756, E 16,41596 ) SMCH ČS 1, 8, 10, 12D, 13, 16, 17, 22B, 26C, 27CH, 27N, 27I, 27J, 27S, 28, 29K, 34C, 35F, 35A, 36B, 37B SMCH KŘ 101A, 102G, 104D, 106L, 107L, 108A, 110A, 111A, 112A, 113D, 114A, 114F, 115A, 116B, 117A, 120H, 121A SM02 SM02/1, SM02/2, SM02/3, SM02/4, SM02/11, SM02/12, SM02/13, SM02/14, SM02/21, SM02/22, SM02/23, SM02/24 SM02/31, SM02/32, SM02/33, SM02/34 * u porostu byla u stromů 1/3 korun proschlých 5
7 Jehlice byly po odběru zamraženy a embryogenní pletiva byla z důvodu vysokého obsahu vody v pletivech lyofilizována. Rostlinný materiál byl rozdrcen na prášek pomocí tekutého dusíku a DNA byla izolována pomocí kitu DNeasy Plant Mini Kit (QIA- GEN) dle dodaného izolačního protokolu (DNeasy Plant Handbook). U získaných vzorků byla měřena koncentrace DNA v ng/µl a její čistota (dána poměrem A260/280) přístrojem Nanophotometer (Implen). Hodnoty získané DNA se pohybovaly v rozmezí ng/µl, eluáty DNA z embryogenních pletiv dosahovaly vyšších hodnot - až 220 ng/µl, čistota se průměrně pohybovala v hodnotách 1,7-1,9, které indikují vysokou čistotu získané DNA (dle manuálu protokolu DNeasy Plant Handbook). Vzorky DNA s vyšší koncentrací byly pro PCR reakce naředěny na hodnotu cca 20 ng/µl. Pro detekci rozdílů v genetické informaci mezi analyzovanými vzorky bylo odzkoušeno několik DNA markerových systémů. Jejich princip je založen na amplifikaci fragmentů (určitých oblastí genomu) technikou PCR polymerázové řetězové reakce. Byly odzkoušeny metody RAPD, PCR RFLP a SSR markery. Obr. 1: Odběry biologického materiálu genetické studie Obr. 2: Odběr vzorků rostlinného materiálu z porostu SMA se sníženou vitalitou stromů RAPD Pro předběžné ověření genetické proměnlivosti sledovaných jedinců smrku ztepilého byla použita metoda odvozená od PCR technik metoda RAPD (Random Amplified Polymorphic DNA polymorfizmus náhodně amplifikované DNA). Jako primer se používá krátký oligonukleotid o délce kolem 10 nukleotidů s libovolnou sekvencí. Při této délce je značná pravděpodobnost, že v genomu bude mnoho míst homologních k použitému oligonukleotidu a některá z nich budou dostatečně blízko u sebe, aby v úseku mezi nimi při využití PCR docházelo k amplifikaci DNA. Tato metoda tedy nevyžaduje znalost cílových sekvencí DNA fragmentů. Výsledkem analýzy je velká skupina amplifikovaných fragmentů, které se pak rozdělí elektroforeticky. Genomy příbuzných, ale často i vzdálenějších druhů jsou velmi shodně organizovány. Většina proužků (záznam amplifikovaných fragmentů po elektroforetickém rozdělení na gelu) bývá shodná, proto je třeba vyhledat primer, který vykazuje různou polohu proužků i u velmi příbuzných organizmů. V naší studii byly testovány primery Operon Biotechnologie GmbH ze sad OPR, OPC a OPBC. PCR reakce probíhala v objemu 25 µl pro jeden vzorek a obsahovala 2,5 µl 10xPCR buffer (100mM Tris- HCl, 6
8 ph = 8,3, 500 mm KCl, 15 mm MgCl 2 ), 2 µl směs dntp (každý 2,5 mm ), 4 µl 10-mer primeru firmy Operon (18 25 pmol), 2 µl DNA a 0,3 µl Takara Taq polymerázy (5 units/µl) (TAKARA Biotechnology, CO., LTD.), doplněno sterilní ultračistou vodou (Sigma, Molecular biology) do objemu 25 µl. RAPD reakce probíhala v termocykleru Perkin Elmer 2400 a Bioer XP cycler. Teplotní režim teplotního cyklovače byl nastaven následovně - 93 C 3 min pro počáteční separaci DNA řetězců, dále se opakovalo 45 cyklů: 92 C 1 min pro denaturaci DNA, 35 C 2 min pro nasedání primerů, 72 C 3 min elongační teplota pro vytváření řetězce, konečná elongační teplota 72 C (10 min) a chlazení 4 C. Amplifikační produkty byly analyzovány elektroforézou na 1,5% agarózových gelech v 0,5% TBE pufru a detekovány barvením ethidiem bromidem (SAMBROOK, RUSSELL 2001). 1,5% agarózový gel se připravil navážením 3 g agarózy (Agarose SER- VA for DNA), která byla ve 200 ml roztoku 0,5% TBE (Tris borate EDTA pufr) zahříváním rozpuštěna. Do vychladlého (ale stále tekutého) čirého roztoku bylo přidáno 10 ml ethidium bromidu. Na gel v horizontální elektroforetické aparatuře se nanášelo 25 ml vzorku vyizolované DNA a 4 ml loading buffer (Sigma). Doba trvání elektroforézy byla 30 minut při napětí 40 V a potom minut při napětí 90 V. Gely byly dokumentovány pod UV zářením pomocí kamerového systému Discovery 10gD. Jako standard pro porovnání velikosti amplifikovaných produktů byl použit 100 bp DNA Ladder (NEW ENGLAND Biolabs). RAPD profily byly vyhodnoceny pomocí specializovaného softwaru UltraQuant. Byl vytipován primer OPBC 17 s nukleotidovou sekvencí CCGTTAGTCC, se kterým byly na gelu získány nejzřetelnější polymorfní profily proužků potvrzující genetickou proměnlivost sledovaných vzorků smrku ztepilého. Nevýhodou RAPD technik je, že výsledky nemusí být při hodnocení populačně genetických parametrů u diploidního materiálu spolehlivé (PERRY et al. 1999). U RAPD markerů převládá dominantní charakter, nelze rozlišit zda je DNA fragment naamplifikovaný z heterozygotního nebo homozygotního lokusu. RAPD markery bývají zřídka kodominantní (WILLIAMS et al. 1990). Dominantní charakter RAPD markerů může zkreslovat výsledky hodnocení genetických parametrů u organismů charakteru diploidů se vzdáleným křížením, k nimž patří i smrk. V případě polymorfního lokusu s recesivní alelou vyskytující se v nízké frekvenci, budou RAPD analýzou tyto lokusy hodnoceny jako monomorfní. Zhodnocení heterozygotů mohou poskytnout např. RFLP markery, které jsou kodominantní. (NKONGOLO 1999). U RAPD metody se také z důvodu krátké délky motivu (tzv. nespecifických primerů) a nižší teploty annealingu (35 C) zvyšuje pravděpodobnost nasednutí primeru na mnoha místech molekuly DNA a mohou se vytvořit i falešné produkty. Výhodou této předběžné metody je rychlost analýz a finanční dostupnost. PCR - RFLP Další ověřovaný DNA marker odvozený od polymerázové řetezové reakce byl PCR - RFLP (Restriction Fragment Lenght Polymorphis polymorfizmus délky restrikčních fragmentů). Metoda spočívá v tom, že po PCR amplifikaci určitého úseku DNA za použití specifických primerů jsou amplifikační produkty štěpeny pomocí restrikčních endonukleáz na fragmenty. Existuje velké množství bakteriálních restrikčních endonukleáz, které se liší tím, že rozpoznávají různé krátké sekvence nukleotidů a štěpí DNA na různě dlouhé fragmenty podle individuálního pořadí bází a podle rozpoznané sekvence. Počet i délka fragmentů je pro daného jedince specifická. Identifikace a analýza produktů štěpení se provádí elektroforetickým dělením. Sekvence specifických primerů dodaných od firmy Invitrogen byla zvolena dle literatury (MAGHULY et al. 2008) a byly odzkoušeny primery pro úseky mitochondriální DNA: mt15 D02 (F - TAT 7
9 CTG ACT TGC CTT ATC, R - ATC CGA ATA CAT ACA CC), mt23 D02 (F - CAC CCT TGC GTA GAC TGG, R - GGT TCA CGC AGT GCT TCT), mt1h01 (F - AAG ATG GAT CGC CCT TAC GC, R - GAG GAG GAG GCT TCG TCG TC). Byly optimalizovány postupy PCR reakce pro dvojici primerů k lokusu mh02 (F - TTT TAG GGC CAT TTG CCT GC, R - TCT ATG GAC AAG AGC CCG ACC T) (JEANDROZ et al. 2002). PCR reakce s polymerázou Takara Taq probíhala v objemu 25 µl pro jeden vzorek a obsahovala 2,5 µl 10xPCR buffer (100 mm Tris - HCl, ph = 8,3, 500 mm KCl, 15 mm MgCl 2 ), 0,5 µl směsi dntps (každý 2,5 mm), specifické primery (forward a reverse 50 µm) každý 0,75 µl (v případě mh02 1 µl), 0,2 µl Takara Taq polymerázy (5 units/µl), 2 µl DNA a reakční směs byla doplněna sterilní ultračistou vodou (Sigma - Aldrich, Molecular biology) do objemu 25 µl. PCR reakce probíhaly v termocyklerech Perkin Elmer 2400 a Bioer XP cycler. Teplotní režim termocykleru byl pro reakce s markery mt15 D02, mt23 D02, mt1h01 nastaven na 94 C 3 min. pro počáteční separaci DNA řetězců, dále se opakovalo v 35 cyklech: 94 C 30 sec. pro denaturaci DNA do jednořetězcových molekul, 52 C 54 C (podle druhu primeru) 3 min. pro nasedání primerů, 72 C 1 min. elongační teplota pro syntézu nových vláken, po těchto cyklech následovala konečná elongační teplota 72 C 10 min. a pak byly testované vzorky chlazeny při 4 C. Pro reakci s markerem mh02 byl teplotní režim nastaven na: 94 C 3 min., dále se opakovalo v 35 cyklech: 94 C 45 sec., 56 C 45 sec., 72 C 2 min., po ukončení cyklů následovalo 72 C 10 min. a nakonec byly testované vzorky chlazeny při 4 C. Amplifikační produkty byly analyzovány elektroforeticky na 1,5% agarózových gelech v 0,5% TBE pufru (Tris borate EDTA pufr, Duchefa Biochemie B.V.) a vizualizovány pomocí ethidium bromidu (SAMBROOK, RUSSELL 2001). Na gel v horizontální elektroforetické aparatuře se nanášelo 5 ml PCR produktu a 5 ml loading buffer (Sigma - Aldrich). Doba trvání elektroforézy byla 30 minut při napětí 40 V a pokračovala 150 minut při napětí 70 V. Gely byly dokumentovány pod UV zářením pomocí kamerového systému Discovery 10gD. Jako standard pro definování velikosti amplifikovaných produktů byl použit 100 bp DNA Ladder (NEW ENGLAND Biolabs). Byly získány amplifikační produkty po jednom bandu od každého vzorku, u markeru mh02 ve velikosti 1000 bp, u markeru mt23-d02 ve velikosti 400 bp, u markeru mt1h01 ve velikosti 700 bp, u markeru mt15-d02 byly detekovány 2 různé alely odpovídající velikosti fragmentů 1100 bp a 1250 bp (obr. 3). Srovnatelný výsledek ve velikosti amplifikovaných fragmentů v lokusu mt15-d02 uvádí MAGHULY et al. (2008) u potomstev smrku ztepilého z kontrolovaného křížení. Dále proběhlo testování různých druhů restrikčních endonukleáz, s cílem získat variabilní restrikční fragmenty. Pro štěpení amplifikovaných produktů byly testovány restrikční endonukleázy EcoRI, HinfI, AluI, MspI, HhaI, FspI, MfeI od New England BioLabs. Vzorky z PCR reakce byly společně s restrikční endonukleázou v reakční směsi s dodaným pufrem a sterilní ultračistou vodou inkubovány 5 hodin při 37 C. Pak byly 15 minut vystaveny teplotě 70 C a po prudkém zchlazení nanášeny do 2% agarózového gelu. Detekce štěpných produktů byla po elektroforéze vizualizována pomocí ethidium bromidu. Jako standard byla použita 100 bp DNA Ladder (NEW ENGLAND Biolabs). Gely byly dokumentovány pod UV zářením pomocí kamerového systému Discovery 10gD. Získané restrikční fragmenty nevykazovaly mezi sledovanými vzorky smrku ztepilého polymorfní charakter. 8
10 Obr. 3: Ukázka záznamu amplifikačních produktů na gelovém nosiči získaných po amplifikaci s primery k markeru mt15-d02, S je označení pro velikostní standard 100 bp DNA Ladder SSR markery V současné době se za vysoce polymorfní DNA markery považují mikrosatelity SSR (simple sequence repeats), proto byly použity ke studiu genetické variability i u námi sledovaných vzorků smrku ztepilého. Mikrosatelity jsou sekvence DNA složené z mnohokrát se opakujících krátkých zpravidla 2-4 báze dlouhých motivů. Mikrosatelitové lokusy patří mezi nejvariabilnější oblasti genomu, polymorfismus je dán zejména rozdílem v počtu opakování základního motivu. Uspořádání motivů v mikrosatelitových lokusech může být jednoduché, složené nebo přerušované. Kodominantní charakter markerů SSR umožňuje rozlišit homozygoty od heterozygotů. Mikrosatelitové lokusy získáme PCR reakcí s primery, které jsou komplementární se sekvencemi sousedícími s mikrosatelitovým lokusem. Sekvence primerů je možné získat prohledáváním zveřejněných sekvencí v různých databázích (např. EMBL, GenBank, DDBJ) nebo v publikacích. Další možností je navržení nových mikrosatelitových lokusů pomocí genomových knihoven, což je postup časově i finančně velmi náročný. Nejprve musí být provedena konstrukce genomové knihovny, dále detekce pozitivních klonů pomocí hybridizace s mikrosatelitovou sondou, sekvenování pozitivních klonů, navržení primerů pro amplifikaci mikrosatelitových lokusů a jejich testování. V genomu smrku ztepilého již byly vyhledávány mikrosatelitové lokusy a k nim navrženy sekvence primerů. SCOTTI et al. (2000a) uvádí, že studium mikrosatelitů je u smrku ztepilého komplikované i z důvodu značné velikosti jeho genomu, který je miliard párů bází dlouhý a obsahuje vysoký počet repetitivních oblastí (PFEIFF- ER et al. 1997), které způsobují tvorbu multilokusů a to komplikuje hodnocení amplifikačních produktů. Pro porovnání velikost lidského genomu je o řád nižší (přes 3 miliardy párů bází). Při genetickém studiu vybraných populací smrku ztepilého jsme vycházeli např. z práce SCOTTI et al. (2000a), který v kódující oblasti cdna vyhledal několik mikrosatelitových lokusů s dinukleotidovou repeticí AG a AC. Protože lokusy byly získány z expresních sekvencí, lze u nich předpokládat, že by nemusely být v genomu tak četné a mohly by poskytovat jednotlivé amplifikační produkty SSR markerů. Autor navrhl pomocí programu PRIMER v.0.5 primery k získaným mikrosatelitovým lokusům, jejich testování proběhlo na smrcích z původních populací Itálie a byly zjištěny sekvence primerů, které poskytovaly amplifikáty. Naším záměrem bylo mikrosatelitové primery otestovat na našich populacích smrku ztepilého, optimalizovat postupy PCR reakcí pro získání zřetelných amplifikačních produktů a vybrat sekvence primerů vedoucích k amplifikaci polymorfních mikrosatelitových lokusů. Z uvedené publikace byly testovány specifické primery pro lokusy PAAC3, PAAC11, PAAC 13, PAAC17, PAAC19 a PAAC23 (tab. 2). Dalšími ověřovanými mikrosatelitovými markery byly SpAG 2 a SpAGD 1, sekvence jejich primerů byly převzaty z práce MELNIKOVA et al. (2012). Reakční směs a teplotní cyklus PCR reakcí byly optimalizovány a modifikovány podle práce YAZDANI et al. (2003). Předběžné hodnocení získaných amplifikačních produktů bylo provedeno po proběhlé elektroforéze na 1,5% agarózových gelech v 0,5% TBE pufru (Tris borate EDTA pufr, Duchefa Biochemie B.V.) a vizualizováno pomocí ethidium bromidu (SAMBROOK, RUSSELL 2001) (obr. 4). 9
11 Obr. 4: Ukázka záznamu amplifikačních produktů na gelovém nosiči získaných s primery k lokusu PAAC23 u vzorků z porostu Fláje - SM3 (velikostní standard 100 bp DNA Ladder je uprostřed amplifikátů) Mezi blízkými genotypy je polymorfismus nižší. Při hodnocení jedinců stejného druhu, je pravděpodobné, že se mohou lišit jen v několika nebo i v jednom opakování motivu (u dinokluotidů to představuje 2 báze) a rozdíly nelze detekovat na agarózovém gelu. Je nutné primery nechat fluorescenčně označit a u získaných amplifikačních produktů provést fragmentační analýzu na genetickém analyzátoru. Hodnotitelné polymorfní fragmenty byly získány s primery k lokusům PAAC19, PAAC23 a SpAGD 1. Tyto primery se nechaly nasyntetizovat fluorescenčně označené s modifikacemi 6FAM, VIC a NED. PCR probíhala pro každý vzorek v celkovém objemu 15 µl s použitím polymerázy Platinum Taq DNA Polymerase (Invitrogen by Life technologies) a s ní dodanými komponenty. Obsahovala 1,5 µl 10xPCR buffer, 2 mm MgCl 2, 0,2 mm směsi dntp, 0,37 jednotek Platinum Taq DNA Polymerase, specifické primery každý 0,1 µm k lokusům PAAC19 a SpAGD 1, 0,2 µm k lokusu PAAC23, ng vyizolované DNA a sterilní ultračistou vodou (Sigma - Aldrich, Molecular biology) byla reakční směs doplněna do objemu 15 µl. PCR reakce probíhaly v termocyklerech Bioer XP cycler, Biorer GenePro a Veriti Thermal cycler. Teplotní režim teplotního cyklovače byl nastaven pro PCR s primery k lokusu PAAC23: 94 C 3 min. (denaturace DNA a aktivace polymerázy), dále se opakovalo v 40 cyklech: 94 C 45 sec. (denaturace DNA do jednořetězcových molekul), 54 C 45 sec. (teplota annealingu přisednutí primeru), 72 C 45 sec. (elongace zabudování volných nukleotidtrifosfátů na základě templátu do nově vznikajicího řetězce). Pak následovala teplota 72 C 20 min. (konečná elongační teplota pro dobudování fragmentů) a nakonec byly testované vzorky chlazeny při 4 C. Pro PCR reakce s primery k lokusům SpAGD 1 a PAAC19 probíhal teplotní režim 94 C 5 min., dále se opakovalo v 37 cyklech: 94 C 45 sec., 57 C 45 sec., 72 C 45 sec. a nakonec následovala 20 min. teplota 72 C. Amplifikační produkty byly před fragmentační analýzou denaturovány. Ke každému vzorku bylo přidáno 11 µl Formamidu (Hi-Di TM Formamide, Applied Biosystem) a 0,4 µl velikostního standardu (Gene ScanTM 600 LIZ Size standard v 2.0, Applied Biosystem) a probíhala inkubace 4 minuty při teplotě 94 C, pak byly vzorky rychle zchlazeny na ledu. Fragmentační analýza byla provedena na genetickém analyzátoru Applied Biosystem Další ověřované mikrosatelitové lokusy WS00716.F13, WS0092.A19, WS0022. B15 pro testování polymorfismu u sledovaných populací smrku ztepilého byly získány na pracovišti ASP (Bavarian office for forest Seeding and Planting) v Teisendorfu (Německo). Tyto markery vyvinul RUN- GIS et al. (2004) z kódující oblasti DNA tzv. EST (expressed sequence tags) - SSR markers (tab. 2). Polymerázová řetězová reakce probíhala s primery těchto lokusů formou multiplexu s primery fluorescenčně označenými 6FAM, VIC, NED. Pro každý vzorek byla reakce provedena v celkovém objemu 15 µl s využitím Type-it Microsatellite PCR Kit (QIAGEN), koncentrace každého primeru byla 0,1 µm a vyizolované DNA bylo ng. PCR reakce probíhala v termocykleru Veriti Thermal cycler (Applied Biosystem). Teplotní režim teplotního cyklovače byl nastaven na 95 C 15 min. (denaturace DNA a aktivace polymerázy), dále se opakovalo v 26 cyklech: 94 C 30 sec. (denaturace DNA do jednořetězcových molekul), 53 C 90 sec. (teplota annealingu přisednutí primeru), 72 C 30 sec. (elongace za- 10
12 budování volných nukleotidtrifosfátů na základě templátu do nově vznikajicího řetězce). Pak následovala teplota 60 C 30 min. (konečná elongační teplota pro dobudování fragmentů) a nakonec byly testované vzorky chlazeny při 4 C. Amplifikační produkty byly před fragmentační analýzou denaturovány stejným postupem jako u předcházejících studovaných mikrosatelitových lokusů. Velikost fragmentů byla odečtena pomocí genetického analyzátoru Applied Biosystem Genetický analyzátor pracuje na principu elektroforetického rozdělení fragmentů DNA v tenké kapiláře naplněné speciálním polymerem. Polymer i zkoumaný vzorek je do kapiláry naplňován automaticky. Detekce sekvenovaných úseků je založena na hodnocení fluorescence z fluorescenčně označených primerů po excitaci argonovým laserem. Přístroj je schopen souběžně detekovat vícebarevnou fluorescenci, což umožňuje v multiplexovém uspořádání hodnotit najednou více markerů. Hodnocení velikosti amplifikačních produktů bylo provedeno pomocí softwarového programu GeneMapper 4.1 (Applied Biosystems), který z výsledku měření velikostního standardu, který je přidáván ke všem vzorkům, stanoví kalibrační křivku a na jejím podkladě ohodnotí velikosti analyzovaných fragmentů. Výstupem programu je elektroforetogram získaných dat ve formátu matice všech spočítaných parametrů. Pro posouzení genetických charakteristik sledovaných populací smrku ztepilého byla získaná data mikrosatelitových markerů statisticky zpracována. Tab. 2: Použité SSR markery a sekvence primerů lokus Forward primer Reverse primer PAAC3 PAAC11 PAAC13 PAAC17 PAAC19 PAAC23 SpAGD 1 SpAG 2 WS00716.F13 WS0092.A19 WS0022.B15 Výsledky CGC TAC CTC AGA TTT CTC CA GTA ATG AAA TGT GTG TGT GTG TGT GT GAT ATT GAT GTA CGC ACT GG GAA ACA AAA ATT ATT ACG CG ATG GGC TCA AGG ATG AAT G TGT GGC CCC ACT TAC TAA TAT CAG GTC AAC CAA CTT GTA AAG CCA GCT CTT CAC GTG TAC TTG ATC TCA AGT AAT GGA CAA ACG ATA CA GAT GTT GCA GGC ATT CAG AG TTT GTA GGT GCT GCA GAG ATG AGA TAT TCC CTC ACA AAG TTG G AGT AGC GAG GGT GGA GAT GG AAT TTT TTG AAC AAG AAG TTT TC ATG CCC TCC TAA TGA ATG AAC TCC AAA CGA TTG ATT TCC CGG GCA TTG GTT TAC AAG AGT TGC ACT TGT TTG GCA TTT TCC C TTC GAA GAT CCT CCA AGA TA TTT CCA ATA GAA TGG TGG ATT T GCA CCA GCA TCG ATT GAC TA TGG CTT TTT ATT CCA GCA AGA Pro předběžné ověření genetické proměnlivosti sledovaných jedinců smrku ztepilého byla použita metoda RAPD a byl vytipován primer OPBC 17 potvrzující genetickou proměnlivost vzorků smrku ztepilého. V případě metody PCR - RFLP (Restriction Fragment Lenght Polymorphis polymorfizmus délky restrikčních fragmentů) nebyl u námi sledovaných vzorků zjištěn polymorfismus. Při použití metody SSR bylo pro testování vzorků smrku ztepilého vyhledáno 6 mikrosatelitových markerů, které vykazovaly polymorfismus a výsledky byly interpretovatelné. Jejich amplifikační produkty dosahovaly velikosti v rozmezí bp u markeru PAAC23, bp u markeru SpAGD 1, bp u markeru PAAC19, bp u markeru WS00716.F13, bp u markeru WS0092.A19, bp u markeru WS0022.B15. Hraniční velikosti alel pro sledované populace jsou uvedeny v tab
13 Tab. 3: Rozsah velikostí alel u hodnocených lokusů pro populace smrku ztepilého Populace smrku ztepilého Lokus PAAC23 Lokus PAAC19 Lokus SpAGD1 Lokus WS00716.F13 Lokus WS0092.A19 Lokus WS0022.B15 SM SM SM SMA SMB SMR SMK SMCH ČS SMCH KŘ SM Výsledky analýz byly zpracovány pomocí statistického programu GenAlEx 6.5 (PEAKALL, SMOUSE 2006, 2012). U hodnocených 200 jedinců z 10 populací bylo celkově detekováno 582 různých polymorfních alel v 6 lokusech, v průměru na populaci 9,7 alel na lokus. Nejvíce polymorfní se jevil lokus SpAGD 1 u kterého bylo v průměru na populaci detekováno 14,8 alel, u lokusu WS00716.F13 to bylo 11,9 alel, u WS0022.B15 10,7 alel, u PAAC19 9,7, u PAAC23 6,6 alel, nejméně polymorfní byl lokus WS0092.A19 u kterého bylo detekováno 4,5 alel. Alelické varianty u sledovaných populací a jednotlivých lokusů jsou uvedeny v tabulce 4. Alelické frekvence u sledovaných populací pro jednotlivé lokusy jsou graficky znázorněné na obrázku 5. Všech 6 lokusů u sledovaných populací bylo 100% polymorfních, na základě vypočítané hodnoty P (procentní podíl polymorfních lokusů). Hodnoty heterozygotnosti, což je podíl heterozygotních jedinců v daném lokusu udává tabulka 5. Pozorovaná heterozygotnost (Ho) u celkového souboru 200 jedinců byla nejvyšší u lokusu WS0022.B15 a to 0,931, očekávaná heterozygotnost (He) byla pro tento lokus nižší 0,826. Průměrné hodnoty heterozygotnosti pro jednotlivé populace jsou uvedeny v tabulce 6. Nejvyšší podíl pozorovaných heterozygotů (hodnota Ho = 0,842) se ukázal v populaci z Flájí označené SM3. Nejnižší hodnoty Ho = 0,668 pak byly získány u populace chlumního ekotypu z Křivoklátska. V tabulce 6 jsou uvedeny i průměrné hodnoty počtu efektivních alel, který byl nejvyšší u populace z Flájí SMA 6,644, tato populace má také nejvyšší počet alelických variant 12,333. Další genetická charakteristika uvedená v tabulce 6 je Shannonův informační index ukazující míru genetické diverzity pro jednotlivé populace. Nejvyšší hodnotu dosáhla populace SMA 1,969 a nejnižší SMR 1,619. Jedna z nejvýznamnějších genetických charakteristik je ohodnocení genetických vzdáleností mezi populacemi, které byly pro sledované vzorky vypočítány na základě Neiovy standardní genetické vzdálenosti (NEI 1972), (tab. 7). Získané hodnoty byly dále zpracovány pomocí analýzy PCoA (Principal Coordinate Analysis analýza hlavních koordinát), která slouží k hodnocení vzdáleností největší variability v datech na základě koeficientů podobnosti mezi jedinci, viz obr. 6. Z PCoA analýzy vyplývá, že geneticky nejblíže jsou si populace z oblasti Krušných hor, více se od nich pouze vzdaluje populace SMR ze Saska. Populace chlumního ekotypu z lokalit Českosaské Švýcarsko a Křivoklátsko jsou seskupeny vzdáleněji od ostatních populací. Geneticky nejvzdálenější se jeví populace SM02 z Orlických hor, která také dosahuje nejvyšších hodnot Neiovy míry ge- 12
14 netických vzdáleností vzhledem k ostatním sledovaným populacím, nejvyšší hodnota byla 0,438 mezi SMR (Sasko) a SM02 (Trčkov Šerlišský kotel - Orlické hory). Hodnoty genetických vzdáleností mezi populacemi jsou vzestupně seřazeny v tabulce 8. Dalším vyhodnoceným genetickým kriteriem byl FST - koeficient Inbreedingu, který porovnává míru genetické diferenciace vzájemně mezi populacemi (tab. 9). Populace jsou párově separovány podle podílu na celkové genetické rozdílnosti. FST koeficient se pohybuje v hodnotách od 0 1 a v případě, že populace jsou v Hardy - Weinbergově rovnováze se stejnou alelickou frekvencí je roven 0. Zjištěné FST párové hodnoty pro hodnocené populace se pohybovaly od 0,011 do 0,067. Což představuje malou až střední genetickou diferenciaci mezi populacemi (WRIGHT 1965). Tab. 4: Alelické varianty u populací smrku ztepilého v jednotlivých lokusech SM1 SM2 SM3 SMA SMB SMK SMR PAAC 23 8,000 6,000 8,000 8,000 6,000 6,000 5,000 6,000 6,000 7,000 PAAC 19 12,000 11,000 11,000 12,000 9,000 7,000 5,000 10,000 9,000 11,000 SpAGD 1 14,000 16,000 17,000 21,000 19,000 14,000 10,000 14,000 10,000 13,000 WS00716.F13 10,000 12,000 14,000 14,000 14,000 12,000 12,000 10,000 10,000 11,000 WS0092.A19 6,000 5,000 4,000 7,000 8,000 3,000 4,000 4,000 2,000 2,000 WS0022.B15 11,000 12,000 11,000 12,000 11,000 9,000 9,000 11,000 11,000 10,000 SMCH ČS SMCH KŘ SM02 Obr. 5: Alelické frekvence u hodnocených populací smrku ztepilého v jednotlivých lokusech Frequency 0,800 0,600 0,400 0,200 0,000 Allele Frequency for PAAC PAAC23 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 Frequency 0,500 0,400 0,300 0,200 0,100 0, Allele Frequency for PAAC PAAC19 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 13
15 Frequency 0,400 0,300 0,200 0,100 0, Allele Frequency for SpAGD SpAGD1 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 Frequency 0,300 0,200 0,100 0,000 Allele Frequency for WS00716.F WS00716.F13 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 Frequency 1,000 0,800 0,600 0,400 0,200 0,000 Allele Frequency for WS0092.A WS0092.A19 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 Frequency 0,500 0,400 0,300 0,200 0,100 0,000 Allele Frequency for WS0022.B WS0022.B15 Locus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCHKŘ SM02 14
16 Tab. 5: Průměrné hodnoty heterozygotnosti u všech sledovaných vzorků pro jednotlivé lokusy Lokus PAAC 23 PAAC 19 SpAGD 1 WS00716.F13 WS0092.A19 WS0022.B15 Ht 0,647 0,851 0,936 0,911 0,443 0,855 Mean He 0,618 0,802 0,884 0,872 0,408 0,826 Mean Ho 0,611 0,889 0,693 0,890 0,444 0,931 Ho = Heterozygotnost pozorovaná He = Heterozygotnost očekávaná Ht = Celková očekávaná heterozygotnost Tab. 6: Průměrné hodnoty statistických charakteristik pro jednotlivé populace ze všech pozorovaných lokusů Populace Na Ne I Ho He SM1 Mean 10,167 5,309 1,785 0,706 0,722 SM2 Mean 10,333 6,171 1,836 0,724 0,730 SM3 Mean 10,833 6,042 1,910 0,842 0,771 SMA Mean 12,333 6,644 1,969 0,715 0,763 SMB Mean 11,167 5,908 1,831 0,706 0,725 SMK Mean 8,500 5,008 1,619 0,744 0,685 SMR Mean 7,500 4,754 1,611 0,790 0,722 SMCH ČS Mean 9,167 4,948 1,721 0,722 0,724 SMCH KŘ Mean 8,000 4,978 1,684 0,668 0,731 SM02 Mean 9,000 5,997 1,844 0,813 0,776 Na = Počet různých alel Ne = Počet efektivních alel I = Shannonův informační index Ho = Heterozygotnost pozorovaná He = Heterozygotnost očekávaná Tab. 7: Genetické vzdálenosti mezi populacemi na základě Neiovy míry genetických vzdáleností SM1 0,000 SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCH KŘ SM02 SM2 0,062 0,000 SM3 0,085 0,079 0,000 SMA 0,075 0,071 0,085 0,000 SMB 0,090 0,069 0,095 0,062 0,000 SMK 0,172 0,138 0,151 0,095 0,164 0,000 SMR 0,202 0,178 0,234 0,163 0,182 0,221 0,000 SMCH ČS 0,151 0,168 0,175 0,104 0,139 0,227 0,280 0,000 SMCH KŘ 0,150 0,178 0,154 0,098 0,161 0,211 0,238 0,107 0,000 SM02 0,285 0,306 0,290 0,287 0,297 0,401 0,438 0,267 0,306 0,000 15
17 Obr. 6: Grafické znázornění genetických vzdáleností sledovaných populací smrku ztepilého Principal Coordinates (PCoA) SMR SM02 Coord. 2 SMK SM2 SM1 SMB SM3 SMA SMCHKŘ SMCH ČS Coord. 1 Tab. 8: Vzestupné seřazení genetických vzdáleností mezi populacemi 1. 0,062 SM1-SM ,164 SMB-SMK 2. 0,062 SMA-SMB 23. 0,168 SM2-SMCH ČS 3. 0,069 SM2-SMB 24. 0,172 SM1-SMK 4. 0,071 SM2-SMA 25. 0,178 SM2-SMR 5. 0,075 SM1-SMA 26. 0,178 SM2-SMCH KŘ 6. 0,079 SM2-SM ,182 SMB-SMR 7. 0,085 SM1-SM ,202 SM1-SMR 8. 0,085 SM3-SMA 29. 0,211 SMK-SMCH KŘ 9. 0,090 SM1-SMB 30. 0,221 SMK-SMR 10. 0,095 SM3-SMB 31. 0,227 SMK-SMCH ČS 11. 0,095 SMA-SMK 32. 0,238 SMR-SMCH KŘ 12. 0,098 SMA-SMCH KŘ 33. 0,267 SMCH ČS-SM ,104 SMA-SMCH ČS 34. 0,280 SMR-SMCH ČS 14. 0,107 SMCH ČS-SMCH KŘ 35. 0,285 SM1-SM ,138 SM2-SMK 36. 0,287 SMA-SM ,139 SMB-SMCH KŘ 37. 0,290 SM3-SM ,150 SM1-SMCH KŘ 38. 0,297 SMB-SM ,151 SM1-SMCH ČS 39. 0,306 SM2-SM ,151 SM3-SMK 40. 0,306 SMCH KŘ-SM ,154 SM3-SMCH KŘ 41. 0,401 SMK-SM ,163 SMA-SMR 42. 0,438 SMR-SM02 16
18 Tab. 9: FST - Koeficient Inbreedingu určující genetickou rozdílnost mezi populacemi Pairwise Population Fst Values SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SM1 0,000 SM2 0,011 0,000 SM3 0,014 0,014 0,000 SMA 0,013 0,013 0,013 0,000 SMB 0,016 0,013 0,017 0,011 0,000 SMK 0,031 0,028 0,029 0,020 0,032 0,000 SMR 0,036 0,032 0,036 0,028 0,031 0,046 0,000 SMCH ČS 0,025 0,028 0,025 0,017 0,023 0,040 0,045 0,000 SMCH KŘ 0,024 0,030 0,022 0,016 0,028 0,037 0,043 0,018 0,000 SM02 0,044 0,047 0,036 0,039 0,044 0,067 0,057 0,038 0,043 0,000 SMCH KŘ SM02 Polymorfní informační obsah (PIC - Polymorphism Information Content ) pro vyjádření genetické variability (stupně polymorfismu) sledovaných populací v hodnocených lokusech (tab. 10) byl vypočítán z hodnot získaných pomocí programu GenAlEx 6.5 (PEAKALL, SMOUSE 2006, 2012). PIC hodnoty pro každý mikrosatelitový lokus byly stanoveny na základě frekvencí alel na lokus pomocí vzorce PIC =1 Σ x i2, kde x i je relativní frekvence i-té alely mikrosatelitového lokusu. Markery byly klasifikovány jako informativní, když hodnota PIC byla 0,5 (SHARMA et al. 2009). Stupeň polymorfismu stanovený hodnotou PIC byl zjištěn nejnižší (0,210) u populace SMK v lokusu WS0092. A19 a nejvyšší (0,928) u populace SMA v lokusu SpAGD 1. Dle průměrných hodnot byly sledované populace nejvariabilnější v lokusu SpAGD 1 (0,884), nejméně variabilní v lokusu WS0092.A19 (0,408). Nejvýraznější rozdíly v míře polymorfismu mezi populacemi byly zjištěny v lokusu WS0092.A19. Průměrné hodnoty polymorfního informačního obsahu souhrnně za všechny lokusy se mezi populacemi výrazně nelišily. Nejvíce polymorfní byla populace SM02 (0,776), nejméně polymorfní SMK (0,685). Největší rozdíl ve variabilitě ve stejném lokusu byl mezi populacemi SMK (0,210) a SMR (0,565) v lokusu WS0092.A19. Tab. 10: Polymorfní informační obsah (PIC) u sledovaných populací pro jednotlivé lokusy. Lokus SM1 SM2 SM3 SMA SMB SMK SMR SMCH ČS SMCH KŘ SM02 PAAC 23 0,611 0,516 0,730 0,650 0,471 0,645 0,556 0,569 0,742 0,693 PAAC 19 0,839 0,867 0,844 0,827 0,784 0,734 0,639 0,789 0,811 0,884 SpAGD 1 0,902 0,910 0,904 0,928 0,920 0,896 0,858 0,837 0,839 0,843 WS00716.F13 0,836 0,890 0,875 0,897 0,896 0,885 0,870 0,862 0,831 0,883 WS0092.A19 0,336 0,375 0,441 0,448 0,485 0,210 0,565 0,422 0,305 0,491 WS0022.B15 0,809 0,823 0,833 0,827 0,794 0,743 0,846 0,867 0,859 0,861 Průměr 0,722 0,730 0,771 0,763 0,725 0,685 0,722 0,724 0,731 0,776 17
19 Diskuse V současnosti mnoho autorů studujících genetickou variabilitou u smrku ztepilého využívá pro analýzy SSR markery. Pro genetický průzkum sledovaných populací smrku ztepilého byly v naší práci použity mikrosatelitové markery (SSR lokusy) vybrané na základě práce RUNGIS et al. (2004), kde autoři pro porovnání genetických charakteristik 23 druhů rodu Picea použili 37 mikrosatelitových markerů. Porovnali jsme velikosti získaných amplifikovaných alel vybraných totožných lokusů pro smrk ztepilý s velikostmi alel uvedených v článku. U lokusu PAAC23 autoři uvádějí velikosti alel 290 a 300 bp (naše výsledky byly bp), u lokusu PAAC19 je uvedena velikost 167 a 175 bp (naše výsledky byly bp), u lokusu WS0092.A a 244 bp (naše výsledky byly ), u lokusu WS0022.B15 je uvedena velikost 210 a 222 (naše výsledky byly bp). Rozpětí velikostí alel u našich vzorků bylo podstatně vyšší vzhledem k tomu, že jsme sledovali 200 jedinců, v rámci námi zjištěného rozpětí se nachází i velikosti alel uvedených v práci Rungise, který uvádí velikosti alel ke každému lokusu zjištěné z analýzy jednoho stromu. V případě existence dvou velikostí alel je hodnocený jedinec smrku ztepilého v uvedených lokusech heterozygotní. Dále autor u vybraných druhů smrků (Picea sitchensis, P. glauca a P. mariana) hodnotil pozorovanou a očekávanou heterozygotnost, hodnocení provedl na souborech o velikosti cca 20 jedinců. U námi sledovaného souboru 200 vzorků si byly hodnoty očekávané a pozorované heterozygotnosti podobnější při srovnání s Rungisem sledovaných malých souborů tří druhů smrků. Genetickou variabilitu pro 6 původních populací z oblasti západních Alp při použití 7 SSR markerů zjišťovali ve své studii MELO- NI et al. (2007). Průměrné hodnoty očekávané heterozygotnosti u sledovaných italských populací se pohybovaly od 0,588 (populace Valdieri) do 0,671 (populace Valsesia). SCOTTI et al. (2000b) analyzoval pomocí SCAR markerů 7 italských populací z oblasti Alp, hodnoty očekávané heterozygotnosti se pohybovaly od 0,358 do 0,468, u námi sledovaných populací se pohybovaly od 0,685 (SMK) do 0,776 (SM02). Ve srovnání s italskými populacemi studovanými výše uvedenými autory vykazovaly námi sledované populace na základě hodnot očekávané heterozygotnosti vyšší genetickou diverzitu. SCHUBERT et al. (2001) zjistil u 110 stromů z bavorské populace smrku ztepilého průměrnou hodnotu pozorované heterozygotnosti 0,635, hodnoty pozorované heterozygotnosti u námi sledovaných populací byly od 0,668 (SM3) do 0,842 (SMCH KŘ), u námi sledovaných populací byla pozorována opět vyšší genetická diverzita. U 18 stromů pocházejících ze 3 italských a 1 švýcarské populace při použití 7 SSR markerů se hodnoty očekávané heterozygotnosti pohybovaly od 0,43 do 0,94 (PFEIFFER et al. 1997). Ke genetické charakterizaci sedmi populací (Rusko, severní Švédsko, jižní Švédsko, Německo, Švýcarsko, Itálie, Rumunsko) HEU- ERTZ et al. (2006) použili FST - koeficient Inbreedingu. Protože porovnávali geograficky vzdálené populace získali vyšší hodnoty FST ve srovnání s populacemi, které jsme hodnotili jen na území ČR. Nejvíce se odlišovala populace z Rumunska, která dosahovala s ostatními populacemi hodnot FST od 0,194 do 0,262. U našich populací jsme získali nejvyšší párovou hodnotu FST 0,067 mezi populacemi SM02 (Trčkov Šerlišský kotel - Orlické hory) a SMK (povodí Rauschenbachu, Sasko). V práci MELONI et al. (2007) byla u sledovaných 6 italských populací nalezena celková hodnota FST 0,05, což naznačuje poměrně nízkou genetickou diverzitu vyplývající ze sledovaných genetických rozdílů mezi populacemi, nejvyšší párová hodnota FST mezi sledovanými populacemi byla 0,089, podobné výsledky uvádí SCOTTI et al. (2006) při použití dinukleotidových, trinukleotidových a chloroplastových SSR markerů. Ve srovnání s výsledky práce MELONI 18
20 et al. (2007) uvádí při použití SCAR markerů u italských populací z oblasti západních Alp SCOTTI et al. (2000b) vyšší úroveň celkové hodnoty F ST (0,118). U 37 populací z oblasti severní Evropy (Norsko, Švédsko, Rusko, Pobaltské republiky) TOLLEFSRUD et al. (2009) zjistili při použití 7 mikrosatelitových markerů nízkou celkovou hodnotu FST (0,029). U námi sledovaných populací se párové hodnoty F ST mezi populacemi pohybovaly od 0,011 do 0,067. Nízká úroveň genetické diverzity mezi populacemi vyjádřená pomocí koeficientu F ST se vyskytuje obecně u lesních dřevin a zvláště u jehličnanů (HAMRICK et al. 1992). TOLLEFSRUD et al. (2008) ve své rozsáhlé studii analyzující 369 evropských populací smrku ztepilého pomocí mitochondriálního markeru zjistili pro populace Českého masivu ve srovnání s populacemi východních Alp a karpatskými populacemi (populace střední Evropy) nízkou genetickou diverzitu (G ST ). U 9 euroasijských populací smrku ztepilého zjištovali MELNIKOVA et al. (2012) genetické parametry i pomocí lokusu SpAGD 1, hodnota očekávané heterozygotnosti byla 0,9 a pozorované heterozygotnosti 0,432 a koeficient inbreedingu - F ST byl 0,145. Námi sledované populace smrku v ČR měly v tomto lokusu hodnotu očekávané heterozygotnosti nižší (0,884) a pozorované heterozygotnosti vyšší (0,693), což by mohlo být způsobeno vyšší frekvencí nulových alel u euroasijských populací. Hodnota F ST (hodnotící genetickou diferenciaci mezi populacemi), která byla dosažena u euroasijských populací byla vyšší, to je nejspíše způsobeno většími geografickými vzdálenostmi mezi euroasijskými populacemi. Hodnotu F ST 0,6 uvádí ve své práci VENDRAMIN et al. (2000) při sledování 97 euroasijských populací smrku ztepilého pomocí chloroplastových mikrosatelitových markerů. Závěr Genetická proměnlivost 10 vybraných porostů smrku ztepilého byla studována pomocí DNA analýz. Pro získání polymorfních lokusů byly testovány DNA markery jako RAPD (Random Amplified Polymorphic DNA), PCR RFLP (Restriction Fragment Lenght Polymorphis) pro mitochondriální úseky DNA a mikrosatelity - SSR (simple sequence repeats). U 6 mikrosatelitových lokusů (PAAC23, PAAC19, SpAGD 1, WS00716.F13, WS0092.A19, WS0022.B15) amplifikační produkty vykazovaly významný polymorfismus, byly interpretovatelné a proto byly užity ke studiu polymorfismu sledovaných smrků. Cílem práce bylo pomocí genetických charakteristik porovnat proměnlivost sledovaných porostů z oblasti povodí nádrží Fláje a Rauschenbach. Pro porovnání byly studovány i dvě populace chlumního ekotypu smrku ztepilého a to z oblasti Českosaského Švýcarska a Křivoklátska a dále jedna populace horského ekotypu z Orlických hor z oblasti Trčkova. Zpracováním molekulárních dat získaných ze souboru 200 jedinců byly získány statistické charakteristiky všech 6 mikrosatelitových markerů. Byly hodnoceny počty a frekvence alel, pozorovaná i očekávaná heterozygotnost, F ST - koeficient Inbreedingu, Shannonův informační index, proběhlo ohodnocení genetických vzdáleností mezi populacemi na základě Neiovy standardní genetické vzdálenosti, na které navazovalo grafické zpracování vzdáleností pomocí analýzy PCoA (Principal Coordinate Analysis), byl kalkulován PIC- polymorfní informační obsah. Z těchto hodnocení vyplývá, že u sledovaných 200 jedinců bylo celkově detekováno 582 různých polymorfních alel v 6 lokusech. Nejvíce alel bylo detekováno u lokusu SpAGD 1, u kterého bylo v průměrně 14,8 alel na populaci. Nejvyšší podíl pozorovaných heterozygotů byl zjištěn v populaci z lokality Fláje SM3. Z hodnocení genetických vzdáleností mezi populacemi a grafického znázornění se ukázalo, že populace z oblasti Krušných hor jsou si geneticky nejblíže a vytvořily samostatnou skupinu, pouze populace SMR ze Saska je od ní vzdálená. 19
21 Dvojice populací chlumního ekotypu z lokalit Českosaské Švýcarsko a Křivoklátsko mají k sobě geneticky nejblíže a jsou vzdálenější od ostatních populací. Geneticky nejvzdálenější se jeví populace SM02 z Orlických hor. Při hodnocení míry genetické diferenciace mezi populacemi pomocí F ST - koeficient Inbreedingu se zjistila malá až střední genetická diferenciace mezi populacemi. Při porovnávání genetické variability sledovaných populací pomocí polymorfního informačního obsahu se ukázalo, že průměrná hodnota míry polymorfismu pro sledované lokusy je u všech hodnocených populací poměrně vysoká a hodnoty polymorfismu se od sebe výrazně neliší. Významné rozdíly v míře polymorfismu byly zjištěny na úrovni jednotlivých lokusů, nejnižší polymorfismus byl v lokusu WS0092.A19 u populace SMK a nejvyšší v lokusu SpAGD 1 u populace SMA. Zhoršený zdravotní stav této populace je vzhledem k poměrně vysoké úrovni polymorfismu pravděpodobně způsoben nepříznivým vlivem stanoviště. Analýzy mikrosatelitových markerů se ukázaly jako vhodné pro detekci genetických charakteristik u studovaných populací smrku ztepilého. Hodnocení variability porostů na sledovaných lokalitách bylo provedeno na základě analýzy 6 mikrosatelitových lokusů. Vybrané lokusy byly ověřeny jako vysoce polymorfní a mohou být využity jako genetické markery pro genetickou analýzu populací smrku ztepilého. Při porovnání genetické variability původních populací smrku ztepilého (horský ekotyp - Orlické hory a chlumní ekotyp Českosaské Švýcarsko a Křivoklátsko) s genetickou variabilitou vyselektované zbytkové populace smrku ztepilého vzniklé umělou obnovou v povodí Rašeliníku, nebyly zjištěny rozdíly v úrovni polymorfismu, což je předpokladem přežití tohoto druhu dřeviny v místních extrémních přírodních podmínkách. Literatura: BROWN T. A Klonování genů a analýza DNA. Z anglického originálu Gene Cloning and DNA Analysis: An Introduction (fifth edition - ISBN- 13: , ISBN-10: ). Vydala Univerzita Palackého v Olomouci. 1. české vydání. Translation Martin Fellner a kol., ISBN HAMRICK J. L., GODT M. J. W., SHERMAN-BROYLES S. L Factors influencing levels of genetic diversity in woody plant species. New Forest, 6: HEUERTZ M., DE PAOLI E., KÄLLMAN T., LARSSON H., JURMAN I., MORGANTE M., LASCOUX M., GYLLENSTRAND N Multilocus Patterns of Nukleotide Diversity, Linkage Disequilibrium and Demographic History of Norway Spruce [Picea abies (L.) Karst]. Genetics, 174: CHMELAŘ J Dendrologie s ekologií lesních dřevin. Část I. Jehličnany. Vysoká škola zemědělská v Brně. JEANDROZ S., BASTIEN D., CHANDELIER A., DU JARDIN P., FAVRE J. M A set of primers for amplification of mitochondrial DNA in Picea abies and other conifer species. Molecular Ecology Notes, 2: MALÁ J., DUJÍČKOVÁ M., KÁLAL J The development of encapsulated somatic embryos of Norway Spruce (Picea abies /L./ Karst.). Communicationes Instituti Forestalis Bohemicae, 18: MAGHULY F., BURG K., PINSKER W., NITTINGER F., PRAZNIK W., FLUCH S Development of Mitochondrial Markers for Population Genetics of Norway Spruce [Picea abies (L.) Karst.]. Silvae Genetica, 57 (1): MELONI M., PERINI D., BINELLI G The distribution of genetic variation in Norway spruce (Picea abies Karst.) populations in the western Alps. Journal of Biogeography, 34: MELNIKOVA M. N., PETROV N. B., LOMOV A. A., LA PORTA N., POLITOV D. V Testing of Microsatellite Primers with Different populations of Eurasian Spruces Picea abies (L.) Karst. and Picea obovata Ledeb. Russian Journal of Genetics, 48: NEI M Genetic distance between populations. American Naturalist, 106: NKONGOLO K. K RAPD variations among pure and hybrid populations of Picea mariana, P. rubens, and P. glauca (Pinaceae), and cytogenetic stability of Picea hybrids: identification of species-specific RAPD markers. Plant Systematics and Evolution, 215:
ÚVOD. Klíčová slova: smrk ztepilý, RAPD, somatická embryogeneze, polymorfizmus Key words: Norway spruce, RAPD, somatic embryogenesis, polymorphism
 In this study, we describe the use of DNA analysis by RAPD method (Random Amplified Polymorphic DNA) to identify a variability of Norway spruce hurst ecotype. Total genomic DNA was isolated by DNA Plant
In this study, we describe the use of DNA analysis by RAPD method (Random Amplified Polymorphic DNA) to identify a variability of Norway spruce hurst ecotype. Total genomic DNA was isolated by DNA Plant
Polymerázová řetězová reakce
 Polymerázová řetězová reakce doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2013 Obsah přednášky 1) Co je to PCR, princip, jednotlivé kroky 2) Technické provedení PCR 3) Fyzikální
Polymerázová řetězová reakce doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2013 Obsah přednášky 1) Co je to PCR, princip, jednotlivé kroky 2) Technické provedení PCR 3) Fyzikální
Využití DNA markerů ve studiu fylogeneze rostlin
 Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
HODNOCENÍ GENETICKÉ DIVERZITY VYBRANÝCH POPULACÍ SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ
 HODNOCENÍ GENEICKÉ DIVERZIY ZPRÁVY VYBRANÝCH LESNICKÉHO POPULACÍ VÝZKUMU, SMRKU 58, ZEPILÉHO 2013 (3): POMOCÍ 273-279 MIKROSAELIOVÝCH MARKERŮ HODNOCENÍ GENEICKÉ DIVERZIY VYBRANÝCH POPULACÍ SMRKU ZEPILÉHO
HODNOCENÍ GENEICKÉ DIVERZIY ZPRÁVY VYBRANÝCH LESNICKÉHO POPULACÍ VÝZKUMU, SMRKU 58, ZEPILÉHO 2013 (3): POMOCÍ 273-279 MIKROSAELIOVÝCH MARKERŮ HODNOCENÍ GENEICKÉ DIVERZIY VYBRANÝCH POPULACÍ SMRKU ZEPILÉHO
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION
 USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
Autoindex nad DNA sekvencemi
 Autoindex nd DNA sekvenemi do. Ing. Jn Holub, Ph.D. ktedr teoretiké informtiky Fkult informčníh tehnologií České vysoké učení tehniké v Prze ENBIK 2014 10. 6. 2014 ENBIK 2014, 10. 5. 2014 J. Holub: Autoindex
Autoindex nd DNA sekvenemi do. Ing. Jn Holub, Ph.D. ktedr teoretiké informtiky Fkult informčníh tehnologií České vysoké učení tehniké v Prze ENBIK 2014 10. 6. 2014 ENBIK 2014, 10. 5. 2014 J. Holub: Autoindex
GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ
 GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE Certifikované METODIKY Certifikovaná metodika Ing. HELENA CVRČKOVÁ, Ph.D Ing. PAVLÍNA MÁCHOVÁ, Ph.D 8/2015 Genetická
GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE Certifikované METODIKY Certifikovaná metodika Ing. HELENA CVRČKOVÁ, Ph.D Ing. PAVLÍNA MÁCHOVÁ, Ph.D 8/2015 Genetická
Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis.
 Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
Mendelova genetika v příkladech. Genetické markery
 Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZTEPILÉHO
 VYUŽIÍ MIKROSAELIOVÝCH ZPRÁVY LESNICKÉHO MARKERŮ PRO VÝZKUMU, HODNOCENÍ 59, 2014 SEMENNÉHO (4): 243-249 SADU SMRKU ZEPILÉHO VYUŽIÍ MIKROSAELIOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZEPILÉHO EVALUAION
VYUŽIÍ MIKROSAELIOVÝCH ZPRÁVY LESNICKÉHO MARKERŮ PRO VÝZKUMU, HODNOCENÍ 59, 2014 SEMENNÉHO (4): 243-249 SADU SMRKU ZEPILÉHO VYUŽIÍ MIKROSAELIOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZEPILÉHO EVALUAION
ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD
 ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD Distinguishing of Wheat Varieties (Tritium aestivum L.) by Method RAPD Zuzana Kohutová, Zuzana Kocourková, Hana Vlastníková, Petr Sedlák
ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD Distinguishing of Wheat Varieties (Tritium aestivum L.) by Method RAPD Zuzana Kohutová, Zuzana Kocourková, Hana Vlastníková, Petr Sedlák
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae).
 Populační studie Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae). American Journal of Botany 87(8): 1128
Populační studie Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae). American Journal of Botany 87(8): 1128
Genetické markery, markery DNA
 Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
Polymerázová řetězová reakce. Základní technika molekulární diagnostiky.
 Polymerázová řetězová reakce Základní technika molekulární diagnostiky. Kdo za to může? Kary Mullis 1983 Nobelova cena 1993 Princip PCR Polymerázová řetězová reakce (polymerase chain reaction PCR) umožňuje
Polymerázová řetězová reakce Základní technika molekulární diagnostiky. Kdo za to může? Kary Mullis 1983 Nobelova cena 1993 Princip PCR Polymerázová řetězová reakce (polymerase chain reaction PCR) umožňuje
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
MOLEKULÁRNÍ BIOLOGIE. 2. Polymerázová řetězová reakce (PCR)
 MOLEKULÁRNÍ BIOLOGIE 2. Polymerázová řetězová reakce (PCR) Náplň praktik 1. Izolace DNA z buněk bukální sliznice - izolační kit MACHEREY-NAGEL 2. PCR polymerázová řetězová reakce (templát gdna) 3. Restrikční
MOLEKULÁRNÍ BIOLOGIE 2. Polymerázová řetězová reakce (PCR) Náplň praktik 1. Izolace DNA z buněk bukální sliznice - izolační kit MACHEREY-NAGEL 2. PCR polymerázová řetězová reakce (templát gdna) 3. Restrikční
ACTIVATION OF DEHYDRIN GENES OF GERMINATE PLANTS OF BARLEY TO DROUGHT AND COLD AKTIVACE DEHYDRINOVÝCH GENŮ KLÍČNÍCH ROSTLIN JEČMENE SUCHEM A CHLADEM
 ACTIVATION OF DEHYDRIN GENES OF GERMINATE PLANTS OF BARLEY TO DROUGHT AND COLD AKTIVACE DEHYDRINOVÝCH GENŮ KLÍČNÍCH ROSTLIN JEČMENE SUCHEM A CHLADEM Dokoupilová Z., Holková L., Chloupek O. Ústav pěstování
ACTIVATION OF DEHYDRIN GENES OF GERMINATE PLANTS OF BARLEY TO DROUGHT AND COLD AKTIVACE DEHYDRINOVÝCH GENŮ KLÍČNÍCH ROSTLIN JEČMENE SUCHEM A CHLADEM Dokoupilová Z., Holková L., Chloupek O. Ústav pěstování
Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK
 MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
LESNICKÝ PRŮVODCE HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ. C er ti f i k ov a n é
 HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr.
HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr.
Využití molekulárních markerů v systematice a populační biologii rostlin. 10. Další metody
 Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU Jednou z nejvhodnějších metod pro detekci minimální reziduální
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU Jednou z nejvhodnějších metod pro detekci minimální reziduální
Genetické markery - princip a využití
 Genetika a šlechtění lesních dřevin Genetické markery - princip a využití Doc. Ing. RNDr. Eva Palátová, PhD. Ing. R. Longauer, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován
Genetika a šlechtění lesních dřevin Genetické markery - princip a využití Doc. Ing. RNDr. Eva Palátová, PhD. Ing. R. Longauer, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován
DETEKCE A IDENTIFIKACE FYTOPATOGENNÍCH BAKTERIÍ METODOU PCR-RFLP
 DETEKCE A IDENTIFIKACE FYTOPATOGENNÍCH BAKTERIÍ METODOU PCR-RFLP Polymerázová řetězová reakce (PCR) je in vitro metoda pro enzymatickou syntézu definované sekvence DNA. Reakce využívá dvou oligonukleotidových
DETEKCE A IDENTIFIKACE FYTOPATOGENNÍCH BAKTERIÍ METODOU PCR-RFLP Polymerázová řetězová reakce (PCR) je in vitro metoda pro enzymatickou syntézu definované sekvence DNA. Reakce využívá dvou oligonukleotidových
Detekce Leidenské mutace
 Detekce Leidenské mutace MOLEKULÁRNÍ BIOLOGIE 3. Restrikční štěpení, elektroforéza + interpretace výsledků Restrikční endonukleasy(restriktasy) bakteriální enzymy štěpící cizorodou dsdna na kratší úseky
Detekce Leidenské mutace MOLEKULÁRNÍ BIOLOGIE 3. Restrikční štěpení, elektroforéza + interpretace výsledků Restrikční endonukleasy(restriktasy) bakteriální enzymy štěpící cizorodou dsdna na kratší úseky
Amplifikační metody v molekulární diagnostice mikroorganismů. doc. RNDr. Milan Bartoš, Ph.D.
 Amplifikační metody v molekulární diagnostice mikroorganismů doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2012 Doporučená literatura 1) Persing et al. (1993): Diagnostic Molecular
Amplifikační metody v molekulární diagnostice mikroorganismů doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2012 Doporučená literatura 1) Persing et al. (1993): Diagnostic Molecular
Hybridizace nukleových kyselin
 Hybridizace nukleových kyselin Tvorba dvouřetězcových hybridů za dvou jednořetězcových a komplementárních molekul Založena na schopnosti denaturace a renaturace DNA. Denaturace DNA oddělení komplementárních
Hybridizace nukleových kyselin Tvorba dvouřetězcových hybridů za dvou jednořetězcových a komplementárních molekul Založena na schopnosti denaturace a renaturace DNA. Denaturace DNA oddělení komplementárních
DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH. Michaela Nesvadbová
 DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH Michaela Nesvadbová Význam identifikace živočišných druhů v krmivu a potravinách povinností každého výrobce je řádně a pravdivě označit
DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH Michaela Nesvadbová Význam identifikace živočišných druhů v krmivu a potravinách povinností každého výrobce je řádně a pravdivě označit
Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/
 Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Genetické markery Genetické markery = znaky, které informují o genotypu - vhodné jsou znaky,
Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Genetické markery Genetické markery = znaky, které informují o genotypu - vhodné jsou znaky,
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR Jednou z nejvhodnějších metod pro detekci minimální reziduální choroby
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR Jednou z nejvhodnějších metod pro detekci minimální reziduální choroby
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII. Martina Nováková, VŠCHT Praha
 MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
Využití molekulárních markerů v systematice a populační biologii rostlin. 12. Shrnutí,
 Využití molekulárních markerů v systematice a populační biologii rostlin 12. Shrnutí, Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length Polymorphism) založené
Využití molekulárních markerů v systematice a populační biologii rostlin 12. Shrnutí, Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length Polymorphism) založené
Molekulární genetika IV zimní semestr 6. výukový týden ( )
 Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika IV zimní semestr 6. výukový týden (5.11. 9.11.2007) Nondisjunkce u Downova syndromu 2 Tři rodokmeny rodin s dětmi postiženými
Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika IV zimní semestr 6. výukový týden (5.11. 9.11.2007) Nondisjunkce u Downova syndromu 2 Tři rodokmeny rodin s dětmi postiženými
Metody studia historie populací. Metody studia historie populací
 1) Metody studia genetické rozmanitosti komplexní fenotypové znaky, molekulární znaky. 2) Mechanizmy evoluce mutace, přírodní výběr, genový posun a genový tok 3) Anageneze x kladogeneze - co je vlastně
1) Metody studia genetické rozmanitosti komplexní fenotypové znaky, molekulární znaky. 2) Mechanizmy evoluce mutace, přírodní výběr, genový posun a genový tok 3) Anageneze x kladogeneze - co je vlastně
Genetická diverzita masného skotu v ČR
 Genetická diverzita masného skotu v ČR Mgr. Jan Říha Výzkumný ústav pro chov skotu, s.r.o. Ing. Irena Vrtková 26. listopadu 2009 Genetická diverzita skotu pojem diverzity Genom skotu 30 chromozomu, genetická
Genetická diverzita masného skotu v ČR Mgr. Jan Říha Výzkumný ústav pro chov skotu, s.r.o. Ing. Irena Vrtková 26. listopadu 2009 Genetická diverzita skotu pojem diverzity Genom skotu 30 chromozomu, genetická
Analýza DNA. Co zjišťujeme u DNA DNA. PCR polymerase chain reaction. Princip PCR PRINCIP METODY PCR
 o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna
 Mendelova genetika v příkladech Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna Roman Longauer, Ústav zakládání a pěstění lesů, MENDELU
Mendelova genetika v příkladech Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna Roman Longauer, Ústav zakládání a pěstění lesů, MENDELU
Jednotné pracovní postupy zkoušení krmiv
 Národní referenční laboratoř Strana KVANTITATIVNÍ STANOVENÍ GENETICKÝCH MODIFIKACÍ METODOU qpcr POMOCÍ ROTOR-GENE PROBE PCR KITU Účel a rozsah Postup slouží ke kvantitativnímu stanovení genetických modifikací
Národní referenční laboratoř Strana KVANTITATIVNÍ STANOVENÍ GENETICKÝCH MODIFIKACÍ METODOU qpcr POMOCÍ ROTOR-GENE PROBE PCR KITU Účel a rozsah Postup slouží ke kvantitativnímu stanovení genetických modifikací
UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC
 UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC VYUŽITÍ DNA MIKROSATELITŮ POUŽÍVANÝCH V PANELU NA URČOVÁNÍ
UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC VYUŽITÍ DNA MIKROSATELITŮ POUŽÍVANÝCH V PANELU NA URČOVÁNÍ
Co zjišťujeme u DNA ACGGTCGACTGCGATGAACTCCC ACGGTCGACTGCGATCAACTCCC ACGGTCGACTGCGATTTGAACTCCC
 Analýza DNA Co zjišťujeme u DNA genetickou podstatu konkrétních proteinů mutace bodové, sekvenční delece/inzerce nukleotidů, chromosomové aberace (numerické, strukturální) polymorfismy konkrétní mutace,
Analýza DNA Co zjišťujeme u DNA genetickou podstatu konkrétních proteinů mutace bodové, sekvenční delece/inzerce nukleotidů, chromosomové aberace (numerické, strukturální) polymorfismy konkrétní mutace,
α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C
 α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C Popis stripů: Pracovní postup Izolace DNA Doporučujeme použít následující kit pro izolaci DNA z plné krve nebo jiných typů vzorků: Spin Micro DNA Extraction
α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C Popis stripů: Pracovní postup Izolace DNA Doporučujeme použít následující kit pro izolaci DNA z plné krve nebo jiných typů vzorků: Spin Micro DNA Extraction
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ
 INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
thaliana. balky. 1. Genetická analýza a identifikace počtu genů 2. Určení DNA markerů v genetické vazbě s genem
 Praktikum z genetiky rostlin JS 2014 Genetická analýza a genetické markery 1. Genetická analýza a identifikace počtu genů odolnosti k padlí u ječmene. 2. Určení DNA markerů v genetické vazbě s genem odolnosti
Praktikum z genetiky rostlin JS 2014 Genetická analýza a genetické markery 1. Genetická analýza a identifikace počtu genů odolnosti k padlí u ječmene. 2. Určení DNA markerů v genetické vazbě s genem odolnosti
ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED
 ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED FREKVENCE ALEL GENU KIT ASOCIOVANÉHO SE STRAKATOSTÍ TOBIANO U KONÍ PLEMENE PAINT HORSE Chalupová P., Déduchová
ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED FREKVENCE ALEL GENU KIT ASOCIOVANÉHO SE STRAKATOSTÍ TOBIANO U KONÍ PLEMENE PAINT HORSE Chalupová P., Déduchová
Využití rep-pcr v bakteriální taxonomii
 Využití rep-pcr v bakteriální taxonomii Pavel Švec Česká sbírka mikroorganismů Přírodovědecká fakulta MU rep-pcr založeny na shlukové analýze PCR produktů získaných s primery komplementárními k rozptýleným
Využití rep-pcr v bakteriální taxonomii Pavel Švec Česká sbírka mikroorganismů Přírodovědecká fakulta MU rep-pcr založeny na shlukové analýze PCR produktů získaných s primery komplementárními k rozptýleným
Havarijní plán PřF UP
 Havarijní plán PřF UP v němž se nakládá s geneticky modifikovanými organismy (GMO), zpracovaný podle 20, odst. 4 zákona č. 78/2004 Sb. pro pracoviště kateder Buněčné biologie a genetiky a Oddělení molekulární
Havarijní plán PřF UP v němž se nakládá s geneticky modifikovanými organismy (GMO), zpracovaný podle 20, odst. 4 zákona č. 78/2004 Sb. pro pracoviště kateder Buněčné biologie a genetiky a Oddělení molekulární
LESNICKÝ PRŮVODCE VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ GENETICKÉ DIVERZITY SMRKU ZTEPILÉHO. C er ti f i k ov a n é
 VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ GENETICKÉ DIVERZITY SMRKU ZTEPILÉHO LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ GENETICKÉ DIVERZITY SMRKU ZTEPILÉHO LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
Metodika analýzy molekulárních markerů u jilmu, Ulmus L.
 Metodika analýzy molekulárních markerů u jilmu, Ulmus L. Metodika byla vypracovaná jako výstup projektu NAZV QI92A247 Charakterizace genetické struktury autochtonních populací jilmů pomocí DNA analýz,
Metodika analýzy molekulárních markerů u jilmu, Ulmus L. Metodika byla vypracovaná jako výstup projektu NAZV QI92A247 Charakterizace genetické struktury autochtonních populací jilmů pomocí DNA analýz,
POLYMERÁZOVÁ ŘETĚZOVÁ REAKCE (PCR)
 POLYMERÁZOVÁ ŘETĚZOVÁ REAKCE (PCR) Polymerázová řetězová reakce (PCR, z anglického Polymerase Chain Reaction) je metoda rychlého zmnožení (amplifikace) vybraného úseku DNA. Množený (amplifikovaný) úsek
POLYMERÁZOVÁ ŘETĚZOVÁ REAKCE (PCR) Polymerázová řetězová reakce (PCR, z anglického Polymerase Chain Reaction) je metoda rychlého zmnožení (amplifikace) vybraného úseku DNA. Množený (amplifikovaný) úsek
ISSR (Inter simple sequence repeat) Jiří Košnar
 ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
MENDELOVA ZEMĚDĚLSKÁ A LESNICKÁ UNIVERZITA V BRNĚ. Agronomická fakulta BAKALÁŘSKÁ PRÁCE
 MENDELOVA ZEMĚDĚLSKÁ A LESNICKÁ UNIVERZITA V BRNĚ Agronomická fakulta BAKALÁŘSKÁ PRÁCE BRNO 2006 Petra PŘICHYSTALOVÁ MENDELOVA ZEMĚDĚLSKÁ A LESNICKÁ UNIVERZITA V BRNĚ Agronomická fakulta Ústav morfologie,
MENDELOVA ZEMĚDĚLSKÁ A LESNICKÁ UNIVERZITA V BRNĚ Agronomická fakulta BAKALÁŘSKÁ PRÁCE BRNO 2006 Petra PŘICHYSTALOVÁ MENDELOVA ZEMĚDĚLSKÁ A LESNICKÁ UNIVERZITA V BRNĚ Agronomická fakulta Ústav morfologie,
Yi TPMT. Diagnostická souprava. Návod k použití. Haasova 27 Brno Česká republika. tel.:
 Yi TPMT Diagnostická souprava Návod k použití Výrobce: YBUX s.r.o. Haasova 27 Brno 616 00 Česká republika IČ 63487951 tel.: +420 541 423 710 e-mail: ybux@ybux.eu Název: Yi TPMT Popis: Diagnostická souprava
Yi TPMT Diagnostická souprava Návod k použití Výrobce: YBUX s.r.o. Haasova 27 Brno 616 00 Česká republika IČ 63487951 tel.: +420 541 423 710 e-mail: ybux@ybux.eu Název: Yi TPMT Popis: Diagnostická souprava
CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION
 CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION DIGITÁLNÍ OBRAZOVÁ ANALÝZA ELEKTROFORETICKÝCH GELŮ *** Vyhodnocování získaných elektroforeogramů: Pro vyhodnocování
CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION DIGITÁLNÍ OBRAZOVÁ ANALÝZA ELEKTROFORETICKÝCH GELŮ *** Vyhodnocování získaných elektroforeogramů: Pro vyhodnocování
Mikrosatelity (STR, SSR, VNTR)
 Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
Analýza DNA. Co zjišťujeme u DNA
 Analýza DNA Co zjišťujeme u DNA Genetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů, záměny), chromosomové aberace (numerické, strukturní) Polymorfismy konkrétní
Analýza DNA Co zjišťujeme u DNA Genetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů, záměny), chromosomové aberace (numerické, strukturní) Polymorfismy konkrétní
Mgr. et Mgr. Lenka Falková. Laboratoř agrogenomiky. Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita
 Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské. doc. RNDr. Ivan Mazura, CSc.
 Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské praxi doc. RNDr. Ivan Mazura, CSc. Historie forenzní genetiky 1985-1986 Alec Jeffreys a satelitní DNA 1980 Ray
Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské praxi doc. RNDr. Ivan Mazura, CSc. Historie forenzní genetiky 1985-1986 Alec Jeffreys a satelitní DNA 1980 Ray
Genetika vzácných druhů zuzmun
 Genetika vzácných druhů Publikace Frankham et al. (2003) Introduction to conservation genetics Časopis Conservation genetics, založeno 2000 (máme online) Objekt studia Genetická diversita Rozložení genetické
Genetika vzácných druhů Publikace Frankham et al. (2003) Introduction to conservation genetics Časopis Conservation genetics, založeno 2000 (máme online) Objekt studia Genetická diversita Rozložení genetické
TECHNIKY PCR. PCR - polymerase chain reaction -polymerázová řetězová reakce
 TECHNIKY PCR PCR - polymerase chain reaction -polymerázová řetězová reakce Přehled Molekulárně-biologický úvod, DNA struktura, replikace, DNA polymerasa Princip procesu PCR Optimalizace PCR Typy PCR Aplikace
TECHNIKY PCR PCR - polymerase chain reaction -polymerázová řetězová reakce Přehled Molekulárně-biologický úvod, DNA struktura, replikace, DNA polymerasa Princip procesu PCR Optimalizace PCR Typy PCR Aplikace
VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS
 VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS OVĚŘENÍ VHODNOSTI VYUŽITÍ PANELU MIKROSATELITŮ PRO URČENÍ PARENTITY VYBRANÝCH PLEMEN PSŮ Bryndová
VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS OVĚŘENÍ VHODNOSTI VYUŽITÍ PANELU MIKROSATELITŮ PRO URČENÍ PARENTITY VYBRANÝCH PLEMEN PSŮ Bryndová
Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae).
 Populační studie Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae). Biological Conservation 109: 249 258.
Populační studie Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae). Biological Conservation 109: 249 258.
CHARACTERISTICS OF WHEAT GENOTYPES USING HIGH MOLECULAR WEIGHT SUBUNITS GLUTENIN ALLELE
 CHARACTERISTICS OF WHEAT GENOTYPES USING HIGH MOLECULAR WEIGHT SUBUNITS GLUTENIN ALLELE CHARAKTERISTIKA GENOTYPŮ PŠENICE POMOCÍ ALEL VYSOKOMOLEKULÁRNÍCH PODJEDNOTEK GLUTENINŮ Kocourková Z., Vejl P. Katedra
CHARACTERISTICS OF WHEAT GENOTYPES USING HIGH MOLECULAR WEIGHT SUBUNITS GLUTENIN ALLELE CHARAKTERISTIKA GENOTYPŮ PŠENICE POMOCÍ ALEL VYSOKOMOLEKULÁRNÍCH PODJEDNOTEK GLUTENINŮ Kocourková Z., Vejl P. Katedra
Molekulární genetika II zimní semestr 4. výukový týden ( )
 Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika II zimní semestr 4. výukový týden (27.10. 31.10.2008) prenatální DNA diagnostika presymptomatická Potvrzení diagnózy Diagnostika
Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika II zimní semestr 4. výukový týden (27.10. 31.10.2008) prenatální DNA diagnostika presymptomatická Potvrzení diagnózy Diagnostika
Inovace studia molekulární a buněčné biologie
 Inovace studia molekulární a buněčné biologie I n v e s t i c e d o r o z v o j e v z d ě l á v á n í reg. č. CZ.1.07/2.2.00/07.0354 Tento projekt je spolufinancován Evropským sociálním fondem a státním
Inovace studia molekulární a buněčné biologie I n v e s t i c e d o r o z v o j e v z d ě l á v á n í reg. č. CZ.1.07/2.2.00/07.0354 Tento projekt je spolufinancován Evropským sociálním fondem a státním
Braf V600E StripAssay
 Braf V600E StripAssay Kat. číslo 5-570 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci čerstvých nebo mražených biopsií použijte soupravy Qiagen QIAmp DNA Mini nebo Micro. Pro izolaci
Braf V600E StripAssay Kat. číslo 5-570 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci čerstvých nebo mražených biopsií použijte soupravy Qiagen QIAmp DNA Mini nebo Micro. Pro izolaci
NRAS StripAssay. Kat. číslo 5-610. 20 testů 2-8 C
 NRAS StripAssay Kat. číslo 5-610 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci DNA musí být použita vhodná metoda vzhledem k typu tkáně vzorku. Pro doporučení vhodné metody kontaktujte
NRAS StripAssay Kat. číslo 5-610 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci DNA musí být použita vhodná metoda vzhledem k typu tkáně vzorku. Pro doporučení vhodné metody kontaktujte
ZAKLÁDÁNÍ SEMENNÝCH SADŮ DRUHÉ
 Lesy České republiky, s. p., Hradec Králové V Ý Z K U M N É P R O J E K T Y GRANTOVÉ SLUŽBY LČR Souhrn projektu ZAKLÁDÁNÍ SEMENNÝCH SADŮ DRUHÉ GENERACE PRO BOROVICI LESNÍ Řešitel Výzkumný ústav lesního
Lesy České republiky, s. p., Hradec Králové V Ý Z K U M N É P R O J E K T Y GRANTOVÉ SLUŽBY LČR Souhrn projektu ZAKLÁDÁNÍ SEMENNÝCH SADŮ DRUHÉ GENERACE PRO BOROVICI LESNÍ Řešitel Výzkumný ústav lesního
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky Populační genetika (KBB/PG)
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky Populační genetika (KBB/PG)
Polymorfismus délky restrikčních fragmentů (RFLP)
 ÚSTAV LÉKAŘSKÉ BIOCHEMIE A LABORATORNÍ DIAGNOSTIKY 1. LF UK Polymorfismus délky restrikčních fragmentů (RFLP) Praktické cvičení z lékařské biochemie Všeobecné lékařství Martin Vejražka 2017/18 Obsah POLYMORFISMUS
ÚSTAV LÉKAŘSKÉ BIOCHEMIE A LABORATORNÍ DIAGNOSTIKY 1. LF UK Polymorfismus délky restrikčních fragmentů (RFLP) Praktické cvičení z lékařské biochemie Všeobecné lékařství Martin Vejražka 2017/18 Obsah POLYMORFISMUS
Crossing-over. over. synaptonemální komplex
 Genetické mapy Crossing-over over v průběhu profáze I meiózy princip rekombinace genetického materiálu mezi maternálním a paternálním chromosomem synaptonemální komplex zlomy a nová spojení chromatinových
Genetické mapy Crossing-over over v průběhu profáze I meiózy princip rekombinace genetického materiálu mezi maternálním a paternálním chromosomem synaptonemální komplex zlomy a nová spojení chromatinových
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin
 Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace praktických cvičení molekulárně-biologických předmětů o sekvenční úlohy PRACOVNÍ PROTOKOL PRO PŘEDMĚT GENETCKÁ DIVERZITA Vypracováno
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace praktických cvičení molekulárně-biologických předmětů o sekvenční úlohy PRACOVNÍ PROTOKOL PRO PŘEDMĚT GENETCKÁ DIVERZITA Vypracováno
LESNICKÝ PRŮVODCE VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ. C er ti f i k ov a n é
 VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. HELENA CVRČKOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. HELENA CVRČKOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE
 STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr. LUCIE POLÁKOVÁ
STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr. LUCIE POLÁKOVÁ
Genotypování: Využití ve šlechtění a určení identity odrůd
 Molekulární přístupy ve šlechtění rostlin Aplikovaná genomika Genotypování: Využití ve šlechtění a určení identity odrůd Miroslav Valárik 14.2. 2017 Šlěchtění rostlin: Cílený výběr a manipulace s genomy
Molekulární přístupy ve šlechtění rostlin Aplikovaná genomika Genotypování: Využití ve šlechtění a určení identity odrůd Miroslav Valárik 14.2. 2017 Šlěchtění rostlin: Cílený výběr a manipulace s genomy
VARIABILITY IN H-FABP, C-MYC, GH, LEP, LEPR GENES IN LARGE WHITE, LANDRACE AND DUROC BREEDS OF PIGS
 VARIABILITY IN H-FABP, C-MYC, GH, LEP, LEPR GENES IN LARGE WHITE, LANDRACE AND DUROC BREEDS OF PIGS VARIABILITA GENŮ H-FABP, C-MYC, GH, LEP, LEPR U PLEMEN PRASAT BÍLÉ UŠLECHTILÉ, LANDRASE A DUROK Mikolášová
VARIABILITY IN H-FABP, C-MYC, GH, LEP, LEPR GENES IN LARGE WHITE, LANDRACE AND DUROC BREEDS OF PIGS VARIABILITA GENŮ H-FABP, C-MYC, GH, LEP, LEPR U PLEMEN PRASAT BÍLÉ UŠLECHTILÉ, LANDRASE A DUROK Mikolášová
ANALYSIS OF SERPINE1 GENE VARIABILITY IN PIGS
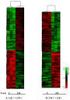 ANALYSIS OF SERPINE1 GENE VARIABILITY IN PIGS Weisz F., Knoll A. Department of Animal Morphology, Physiology and Genetics, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
ANALYSIS OF SERPINE1 GENE VARIABILITY IN PIGS Weisz F., Knoll A. Department of Animal Morphology, Physiology and Genetics, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Poziční klonování Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s metodou pozičního klonování genů
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Poziční klonování Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s metodou pozičního klonování genů
Využití molekulárních markerů v systematice a populační biologii rostlin. 2. Přehled aplikací a otázek
 Využití molekulárních markerů v systematice a populační biologii rostlin 2. Přehled aplikací a otázek Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length
Využití molekulárních markerů v systematice a populační biologii rostlin 2. Přehled aplikací a otázek Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length
Bi5130 Základy práce s lidskou adna
 Bi5130 Základy práce s lidskou adna Mgr. et Mgr. Kristýna Brzobohatá pizova@sci.muni.cz Laboratoř biologické a molekulární antropologie, ÚEB, PřF, Mu Bi5130 Základy práce s lidskou adna PCR polymerase
Bi5130 Základy práce s lidskou adna Mgr. et Mgr. Kristýna Brzobohatá pizova@sci.muni.cz Laboratoř biologické a molekulární antropologie, ÚEB, PřF, Mu Bi5130 Základy práce s lidskou adna PCR polymerase
EGFR XL StripAssay. Kat. číslo 5-630. 20 testů 2-8 C
 EGFR XL StripAssay Kat. číslo 5-630 20 testů 2-8 C Popis stripu Pracovní postup 1. Izolace DNA Pro izolaci DNA použijte vhodný izolační kit. Doporučené kity jsou následující: Pro izolaci čerstvých nebo
EGFR XL StripAssay Kat. číslo 5-630 20 testů 2-8 C Popis stripu Pracovní postup 1. Izolace DNA Pro izolaci DNA použijte vhodný izolační kit. Doporučené kity jsou následující: Pro izolaci čerstvých nebo
Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO.
 Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO. PCR Nucleotide Mix Předem připravený roztok ultračistých PCR deoxynukleotidů (datp,
Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO. PCR Nucleotide Mix Předem připravený roztok ultračistých PCR deoxynukleotidů (datp,
Genetický polymorfismus
 Genetický polymorfismus Za geneticky polymorfní je považován znak s nejméně dvěma geneticky podmíněnými variantami v jedné populaci, které se nachází v takových frekvencích, že i zřídkavá má frekvenci
Genetický polymorfismus Za geneticky polymorfní je považován znak s nejméně dvěma geneticky podmíněnými variantami v jedné populaci, které se nachází v takových frekvencích, že i zřídkavá má frekvenci
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
CYCLER CHECK. Test pro validaci teplotní uniformity cyklérů. Připraveno k použití, prealiquotováno. REF 7104 (10 testů) REF (4 testy)
 CZ Návod k použití CYCLER CHECK Test pro validaci teplotní uniformity cyklérů Připraveno k použití, prealiquotováno REF 7104 (10 testů) REF 71044 (4 testy) Obsah 1. Popis výrobku... 2 2. Materiál... 3
CZ Návod k použití CYCLER CHECK Test pro validaci teplotní uniformity cyklérů Připraveno k použití, prealiquotováno REF 7104 (10 testů) REF 71044 (4 testy) Obsah 1. Popis výrobku... 2 2. Materiál... 3
Optimalizace metody PCR pro její využití na vzorky KONTAMINOVANÝCH PITNÝCH VOD
 Optimalizace metody PCR pro její využití na vzorky KONTAMINOVANÝCH PITNÝCH VOD Dana Vejmelková, Milan Šída, Kateřina Jarošová, Jana Říhová Ambrožová VODÁRENSKÁ BIOLOGIE, 1. 2. 2017 ÚVOD Sledované parametry,
Optimalizace metody PCR pro její využití na vzorky KONTAMINOVANÝCH PITNÝCH VOD Dana Vejmelková, Milan Šída, Kateřina Jarošová, Jana Říhová Ambrožová VODÁRENSKÁ BIOLOGIE, 1. 2. 2017 ÚVOD Sledované parametry,
V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014. Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU
 V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU Zemědělská 1, Budova A, 4. patro (učebny dle programu)
V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU Zemědělská 1, Budova A, 4. patro (učebny dle programu)
AFLP. protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech
 AFLP AFLP protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech AFLP protokoly kit Invitrogen bez použití kitu AmpliTaq Gold (ABI) možnost
AFLP AFLP protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech AFLP protokoly kit Invitrogen bez použití kitu AmpliTaq Gold (ABI) možnost
Co zjišťujeme u DNA ACGGTCGACTGCGATGAACTCCC ACGGTCGACTGCGATCAACTCCC ACGGTCGACTGCGATTTGAACTCCC
 Analýza DNA Co zjišťujeme u DNA Genetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní
Analýza DNA Co zjišťujeme u DNA Genetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní
2 Inkompatibilita v systému Rhesus. Upraveno z A.D.A.M.'s health encyclopedia
 2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
Metody používané v MB. analýza proteinů, nukleových kyselin
 Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci. reg. č.: CZ.1.07/2.2.00/
 Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci reg. č.: CZ.1.07/2.2.00/28.0088 Hybridizační metody v diagnostice Mgr. Gabriela Kořínková, Ph.D. Laboratoř molekulární
Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci reg. č.: CZ.1.07/2.2.00/28.0088 Hybridizační metody v diagnostice Mgr. Gabriela Kořínková, Ph.D. Laboratoř molekulární
6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života?
 6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života? Pamatujete na to, co se objevilo v pracích Charlese Darwina a Alfreda Wallace ohledně vývoje druhů? Aby mohl mechanismus přírodního
6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života? Pamatujete na to, co se objevilo v pracích Charlese Darwina a Alfreda Wallace ohledně vývoje druhů? Aby mohl mechanismus přírodního
Jaroslava Ovesná, Jan Hodek, Lucie Pavlátová,
 Jaroslava Ovesná, Jan Hodek, Lucie Pavlátová, METODIKA DETEKCE GENETICKY MODIFIKOVANÉHO TABÁKU VIRŽINSKÉHO (Nicotina tabacum L. cv. Samsun) S VNESENÝM KVASINKOVÝM MITOTICKÝM AKTIVÁTOREM (gen SpCdc25 z
Jaroslava Ovesná, Jan Hodek, Lucie Pavlátová, METODIKA DETEKCE GENETICKY MODIFIKOVANÉHO TABÁKU VIRŽINSKÉHO (Nicotina tabacum L. cv. Samsun) S VNESENÝM KVASINKOVÝM MITOTICKÝM AKTIVÁTOREM (gen SpCdc25 z
VARIABILITY OF THE PORCINE MYOD1 GENE VARIABILITA GENU MYOD1 U PRASAT
 Verner J., Kuciel J. VARIABILITY OF THE PORCINE MYOD1 GENE VARIABILITA GENU MYOD1 U PRASAT Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická univerzita v Brně, Zemědělská 1, 613 00,
Verner J., Kuciel J. VARIABILITY OF THE PORCINE MYOD1 GENE VARIABILITA GENU MYOD1 U PRASAT Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická univerzita v Brně, Zemědělská 1, 613 00,
Genové knihovny a analýza genomu
 Genové knihovny a analýza genomu Klonování genů Problém: genom organismů je komplexní a je proto obtížné v něm najít a klonovat specifický gen Klonování genů Po restrikčním štěpení genomové DNA pocházející
Genové knihovny a analýza genomu Klonování genů Problém: genom organismů je komplexní a je proto obtížné v něm najít a klonovat specifický gen Klonování genů Po restrikčním štěpení genomové DNA pocházející
"Učení nás bude více bavit aneb moderní výuka oboru lesnictví prostřednictvím ICT ". Základy genetiky, základní pojmy
 "Učení nás bude více bavit aneb moderní výuka oboru lesnictví prostřednictvím ICT ". Základy genetiky, základní pojmy 1/75 Genetika = věda o dědičnosti Studuje biologickou informaci. Organizmy uchovávají,
"Učení nás bude více bavit aneb moderní výuka oboru lesnictví prostřednictvím ICT ". Základy genetiky, základní pojmy 1/75 Genetika = věda o dědičnosti Studuje biologickou informaci. Organizmy uchovávají,
Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/
 Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 ZÁKLADNÍ GENETICKÉ POJMY Genetika je nauka o dědičnosti a proměnlivosti znaků. Znakem se
Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 ZÁKLADNÍ GENETICKÉ POJMY Genetika je nauka o dědičnosti a proměnlivosti znaků. Znakem se
Jan Hodek, Jaroslava Ovesná, Lucie Pavlátová. METODIKA DETEKCE GENETICKY MODIFIKOVANÉ PAPÁJI LINIÍ 55-1 a 63-1 METODIKA PRO PRAXI
 Jan Hodek, Jaroslava Ovesná, Lucie Pavlátová METODIKA DETEKCE GENETICKY MODIFIKOVANÉ PAPÁJI LINIÍ 55-1 a 63-1 METODIKA PRO PRAXI Výzkumný ústav rostlinné výroby, v.v.i. 2008 Metodika byla vypracována pracovníky
Jan Hodek, Jaroslava Ovesná, Lucie Pavlátová METODIKA DETEKCE GENETICKY MODIFIKOVANÉ PAPÁJI LINIÍ 55-1 a 63-1 METODIKA PRO PRAXI Výzkumný ústav rostlinné výroby, v.v.i. 2008 Metodika byla vypracována pracovníky
Pokročilé biofyzikální metody v experimentální biologii
 Pokročilé biofyzikální metody v experimentální biologii Ctirad Hofr 1/1 Proč biofyzikální metody? Biofyzikální metody využívají fyzikální principy ke studiu biologických systémů Poskytují kvantitativní
Pokročilé biofyzikální metody v experimentální biologii Ctirad Hofr 1/1 Proč biofyzikální metody? Biofyzikální metody využívají fyzikální principy ke studiu biologických systémů Poskytují kvantitativní
Metody používané v MB. analýza proteinů, nukleových kyselin
 Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
