LESNICKÝ PRŮVODCE VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ GENETICKÉ DIVERZITY SMRKU ZTEPILÉHO. C er ti f i k ov a n é
|
|
|
- Filip Vítek
- před 5 lety
- Počet zobrazení:
Transkript
1 VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ GENETICKÉ DIVERZITY SMRKU ZTEPILÉHO LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. OLGA TRČKOVÁ 6/2018
2 Využití mikrosatelitových markerů pro hodnocení genetické diverzity smrku ztepilého Certifikovaná metodika Ing. Helena Cvrčková, Ph.D. Ing. Pavlína Máchová, Ph.D. Ing. Olga Trčková Strnady
3 Lesnický průvodce 6/2018 Výzkumný ústav lesního hospodářství a myslivosti, v. v. i. Strnady 136, Jíloviště Publikace vydané v řadě Lesnický průvodce jsou dostupné v elektronické verzi na: Vedoucí redaktor: Ing. Jan Řezáč; rezac@vulhm.cz Výkonná redaktorka: Miroslava Valentová; valentova@vulhmop.cz Grafická úprava a zlom: Klára Šimerová; simerova@vulhm.cz ISBN ISSN
4 USE OF MICROSATELLITE MARKERS FOR EVALUATING GENETIC DIVERSITY OF NORWAY SPRUCE Abstract The aim of this methodology is to present the use of DNA analyses by nuclear microsatellite markers for genetic characterization and clonal identification of Picea abies (L.) Karsten. The methodology describes the processes of sampling, isolation of DNA, conditions of the polymerase chain reaction (PCR), separation and sizing of amplification products, and molecular data calculations. Twelve selected variable microsatellite markers were proved suitable for finding the genetic parameters of Norway spruce stands and seven high polymorphic nuclear microsatellite markers were selected for verifying the clonal identity. Knowledge based on DNA analyses regarding the variability of genetic resources will contribute to the quality of the reproduction material and the creation of optimal species composition in forests. Key words: Norway spruce; microsatellites; DNA analysis; genetic diversity; genetic differentiation; clonal identification Oponenti: Ing. Alžběta Pařízková, ÚHÚL, Brandýs n. L., pobočka Hradec Králové RNDr. Slavomír Rakouský, CSc., odborný konzultant, České Budějovice 3
5 Foto na obálce: Autorské právo: Adresy autorů: Ing. Helena Cvrčková, Ph.D. Ing. Pavlína Máchová, Ph.D. Ing. Olga Trčková Výzkumný ústav lesního hospodářství a myslivosti, v. v. i. Strnady 136 Jíloviště cvrckova@vulhm.cz machova@vulhm.cz trckova@vulhm.cz
6 Obsah: I CÍL METODIKY... 7 II VLASTNÍ POPIS METODIKY ÚVOD METODICKÉ POSTUPY... 9 a Odběr vzorků a postupy izolace DNA... 9 b Postupy PCR amplifikace c Postupy elektroforézy v agarózovém gelu d Postupy fragmentační analýzy a hodnocení PCR produktů e Zpracování molekulárních dat III SROVNÁNÍ NOVOSTI POSTUPŮ IV POPIS UPLATNĚNÍ CERTIFIKOVANÉ METODIKY V EKONOMICKÉ ASPEKTY VI DEDIKACE VII SEZNAM POUŽITÉ SOUVISEJÍCÍ LITERATURY VIII SEZNAM PUBLIKACÍ, KTERÉ PŘEDCHÁZELY METODICE Summary Příloha... 39
7 Zkratky použité v textu: DNA deoxyribonukleová kyselina nssr nuclear Simple Sequence Repeats (jaderné mikrosatelity) SSR Simple Sequence Repeats (mikrosatelity) PCR Polymerase Chain Reaction (polymerázová řetězová reakce)
8 I CÍL METODIKY Cílem metodiky je představit postupy analýz DNA s využitím rozšířeného spektra jaderných mikrosatelitových lokusů pro zhodnocení úrovně genetické diverzity a posouzení rozdílů genetických struktur u významných porostů a populací smrku ztepilého za účelem zachování stability lesních ekosystémů. A dále představit objektivní postupy ověřování deklarované identity klonů s pomocí vysoce polymorfních mikrosatelitových markerů. II VLASTNÍ POPIS METODIKY 1 Úvod Smrk ztepilý (Picea abies (L.) Karsten) zařazený do čeledi Pinacea je z hlediska hospodářského využití nejvýznamnějším druhem jehličnaté dřeviny střední a severní Evropy. V první polovině 19. století se svým rychlým růstem a hlavně technickými přednostmi dřeva stal hlavní hospodářskou dřevinou, která ovládla náš dřevařský trh (Svoboda 1953). V posledních desetiletích bylo lesnickým záměrem omezit smrkové porosty a dosáhnout vyvážené proporce mezi listnatými a jehličnatými dřevinami za účelem zachování druhově vyvážených zdravých lesních porostů. Vlivem dramatických klimatických změn posledních let především z nedostatku vláhy naopak začalo docházet k hromadnému napadání suchem oslabeného smrku lýkožroutem smrkovým a především na území Moravy vznikají kalamitní holiny. Smrk ztepilý je dřevina velkých rozměrů, s relativně mělkou povrchovou kořenovou soustavou, značně náročná na půdní vlhkost a vyžaduje i vyšší relativní vlhkost vzduchu. Citlivější je k vysokým teplotám a na znečištění ovzduší. Nedostatek vláhy je limitujícím faktorem jeho dobrého růstu (Úradníček et al. 2009). Pro zachování genových zdrojů lesních porostů, musí probíhat v dostatečné míře přirozená regenerace, která souvisí s adaptačním potenciálem původních stromů (Nowakowska et al. 2014). Obecně se předpokládá, že populace vyznačující se nízkou genetickou rozmanitostí, by mohly být podstatně citlivější na změny životního prostředí, napadení chorobami a škůdci (Maghuly et al. 2006). Genetická rozmanitost a biodiverzita populací lesních dřevin je zásadní podmínkou pro přizpůsobení se změnám klimatu (Hampe et Petit 2005; Neale et al. 2011) a zajištění stability 7
9 lesních ekosystémů (Whitham et al. 2006; Nowakowska et al. 2014). Znalosti založené na analýze DNA, pokud jde o ověření variability genetických zdrojů, přispějí ke kvalitě reprodukčního materiálu. U smrku ztepilého bylo již provedeno mnoho genetických studií pomocí izoenzymů (Geburek 1999; Konnert 2009) a analýz jaderných, mitochondriálních a chloroplastových DNA markerů (Pfeiffer et al. 1997; Vendramin et al. 2000; Schubert et al. 2001; Bozhko et al. 2003; Maghuly et al. 2008; Tollefsrud et al. 2009; Unger et al. 2011; Cvjetković et al. 2017). Účelem zde zpracovávaných metodických postupů bylo pro ověřování genetické rozmanitosti smrku ztepilého vybrat vhodné jaderné mikrosatelity, nuclear simple sequence repeats (nssr) markery. Mikrosatelitové markery se široce využívají pro hodnocení genetické diverzity populací lesních dřevin. Mikrosatelity jsou složené z mnohokrát se opakujících krátkých motivů nukleotidů zpravidla 2 4 báze dlouhých a jejich polymorfismus je dán zejména rozdílem v počtu opakování základního motivu nukleotidů. Pro hodnocení jejich délky musíme získat milióny jejich kopií pomocí polymerázové řetězové reakce (PCR). PCR probíhá s primery, které jsou komplementární se sekvencemi sousedícími s mikrosatelitovým lokusem a její postupy je nutné zoptimalizovat, abychom získávali jednoznačné a reprodukovatelné amplifikáty. Velikosti alel sledovaných lokusů se statisticky zpracovávají pro získání genetických charakteristik šetřených populací smrku ztepilého. Mikrosatelitové markery pro studium genetické struktury a diverzity smrkových porostů již uplatnila řada autorů (Scotti et al. 2000, 2006; Rungis et al. 2004; Meloni et al. 2007; Tollefsrud et al. 2009; Fluch et al. 2011; Unger et al. 2011; Nowakowska et al. 2014; Cvjetković et al. 2017; Verbylaitè et al. 2017). Využití polymorfních mikrosatelitových markerů je významné i pro ověřování deklarované klonové identity zdrojů reprodukčního materiálu například v semenných sadech, klonových archivech atd. Genetické otestování deklarovaného původu reprodukčního materiálu umožňuje objektivní kontrolou potvrdit nebo vyvrátit klonovou identitu ramet. Aplikace nových kontrolních metod klonové identity zdrojů reprodukčního materiálu by měla státní správě vytvořit předpoklady pro formulaci dotační politiky v oblasti podpory zachování a reprodukce genofondu lesních dřevin. 8
10 2 Metodické postupy a Odběr vzorků a postupy izolace DNA Nejvhodnější období pro odběr rostlinného materiálu je jaro nebo počátek léta, kdy lze odebírat pupeny či mladé jehlice. Starší jehlice lze také použít, ale z důvodu vyššího obsahu fenolických látek a polysacharachidů se snižuje kvalita i kvantita vyizolované DNA, což může zkomplikovat průběh navazující PCR pro získání jednoznačných a reprodukovatelných amplifikovaných lokusů. Při zpracování většího počtu vzorků je lépe odebírat mladé jehlice, zpracování pupenů je časově náročnější. Při manipulaci se vzorky je nutná evidenční kontrola, aby nedošlo k záměně mezi vzorky. Vzorky se tedy při odběru označí, uloží do mikroténového sáčku a udržují se při nízké teplotě (chladové tašky s namraženými destičkami) a co nejrychleji se přepraví ke zpracování v laboratoři. Vzhledem k následným analýzám je důležité vzorky držet stále při nízké teplotě. DNA lze izolovat okamžitě z takto čerstvě odebraných vzorků. V případě, že izolace DNA není provedena ihned po přijetí vzorků je nutné vzorky po převedení do laboratorního režimu (zaevidování, nastřihání na vhodnou velikost apod.) uložit minimálně do -20 C. Další možností jak uchovat vzorky je jejich vysušení za pomocí lyofilizátoru a poté je vzduchotěsně uzavřít v nádobách (lze použít plastové falkonky, scintilační lahvičky s dobře těsnícím uzávěrem). Takto zpracovaný (lyofilizovaný) materiál je snadněji zpracovatelný při následném tření vzorků, odpadá nutnost držet homogenizované vzorky na ledu. Pro izolaci DNA u lesních dřevin se nejlépe osvědčila metoda využívající soupravu DNeasy Plant Mini Kit od firmy QIAGEN dle dodaného protokolu (Quick-Start Protocol). Před zahájením vlastního postupu izolace se přidá ethanol ke koncentrátům pufrů AW1 a AW2. Inkubační lázeň se nechá nahřívat na 65 C. V případě výskytu sraženin v pufrech AP1 a AW1 se roztoky nahřejí pro odstranění těchto sraženin. Protokol izolace DNA z rostlinných pletiv s použitím Dneasy Plant Mini Kitu: 1. Maximální množství výchozího čerstvého rostlinného pletiva je 100 mg, v případě lyofilizovaného pletiva 20 mg, rostlinné pletivo je potřeba rozdrtit na prášek, například za použití tekutého dusíku aplikovaného na navážený rostlinný materiál v třecích miskách. Utřený materiál se přenese do 1,5ml mikrocentrifugační zkumavky. 2. K rozdrcenému vzorku se napipetuje 400 ml pufru AP1 a následně 4 ml RnázyA, pomocí vortexu je nutné obsah důkladně protřepat. Získaná směs se ne- 9
11 chá inkubovat 10 minut při 65 C, během inkubace se musí směs promíchávat 2 3 převracením zkumavek. 3. Přidá se 130 ml pufru P3, krátce se promíchá pomocí vortexu a inkubuje 5 minut na ledu a poté centrifuguje 5 minut při rychlosti rpm. 4. Vzniklý lyzát se přepipetuje do QIAshredder Mini Spin kolonky umístěné v 2ml zkumavce (collection tube) a centrifuguje 2 minuty při rychlosti otáček za minutu (rpm). 5. Přefiltrovaná frakce se přepipetuje do nové 1,5ml mikrocentrifugační zkumavky s odečtením získaného objemu. Dáváme pozor, abychom nenabrali případný pelet. 6. Přidáme pufr AW1 v množství odpovídajícímu 1,5násobku objemu odebrané frakce a ihned opakovaným nasáváním a vypouštěním z mikropipety vzniklou směs promícháme. 7. Odpipetujeme 650 ml směsi a přemístíme do Dneasy Mini spin kolonky umístěné v 2ml zkumavce (collection tube) a centrifugujeme 1 minutu při rpm, přefiltrovanou kapalinu odstraníme. Opakujeme tento krok se zbytkem vzorku. 8. Dneasy Mini spin kolonku umístíme do nové 2ml zkumavky, přidáme 500 ml pufru AW2 a centrifugujeme 1 minutu při rpm, přefiltrovanou kapalinu odstraníme. 9. Přidáme dalších 500 ml pufru AW2 a centrifugujeme 2 minuty při rychlosti rpm. Poté vyndáme opatrně Dneasy Mini spin kolonku ze zkumavky, abychom se nedotkli proteklé kapaliny. 10. Přeneseme Dneasy Mini spin kolonku do nové 1,5ml mikrocentrifugační zkumavky. 11. Přidáme 100 ml AE pufru, který aplikujeme přímo na membránu Dneasy Mini spin kolonky. Necháme inkubovat 5 minut při pokojové teplotě a poté centrifugujeme 1 minutu při rychlosti rpm. 12. Opakujeme krok dle bodu 11 pro získání druhého eluátu. 10
12 Požadované přístrojové a materiálové vybavení: analytické váhy, digitální suchá lázeň, centrifuga, vortex, chladicí blok na mikrozkumavky (- 20 C LABTOP COOLERS), mrazicí box, sada pipet a příslušné (sterilní) špičky, sterilní mikrozkumavky Epppendorf 1,5 ml s víčky, sušička nebo sterilizátor U vyizolované DNA lze zjistit její koncentraci v ng/µl a čistotu (na základě poměru absorbancí při 260 nm a 280 nm) přístrojem Nanophotometer (Implen). Koncentrace DNA se pohybovaly v rozmezí ng/µl z mladých jehlic, z pupenů dosahovaly hodnot i nad 300 ng/µl. Hodnoty optimální čistoty by se měly pohybovat v rozmezí 1,7 1,9, nižší nebo vyšší hodnoty indikují přítomnost dalších látek (proteinů, fenolických látek). Kvalita DNA je podstatná pro získání požadovaných PCR amplifikátů. Optimální hodnoty DNA koncentrace dosahují ng/µl. Vzorky DNA s vyšší koncentrací je pro optimální průběh PCR vhodné naředit. b Postupy PCR amplifikace Pro DNA analýzy byla zvolena metoda nuclear simple sequence repeats (nssr) jaderné mikrosatelity. Z testovaných mikrosatelitových markerů bylo pro získání genetických charakteristik vybráno 12 dostatečně polymorfních jaderných mikrosatelitových lokusů PAAC23, PAAC19 z publikace Scotti et al. (2000), EATC1B02 z publikace Scotti et al. (2002), SpAGD 1, SpAGG 3, SpAGC 2, SpAG 2 z publikace Melnikova et al. (2012), WS00716.F13, WS0022.B15, WS0073.H08, WS K13 a WS0023.B03 z publikace Rungis et al. (2004), viz Tab. 1. Kromě markeru EATC1B02 s třínukleotidovu repeticí mají ostatní markery charakter dvounukleotidových motivů. Markery PAAC23, PAAC19, EATC1B02, WS00716.F13, WS0022. B15, WS0073.H08, WS00111.K13, WS0023.B03 byly odvozeny z kódující oblasti jaderného genomu (expressed sequence tags simple sequence repeats). Pro získání amplifikačních produktů těchto lokusů byly optimalizovány reakční směsi a teplotní cykly polymerázové řetězové reakce (PCR) a vypracovány protokoly. PCR s vybranými 12 markery byly sestaveny do čtyř multiplexů. Reakční směsi je nutné připravovat na ledu nebo chladové destičce, DNA polymerázu dáváme do směsi nakonec přímo z mrazicího boxu. 11
13 Tab. 1: Vybrané mikrosatelitové lokusy a sekvence primerů Lokusy Sekvence primerů (5-3 ) PAAC23 PAAC19 SpAGD 1 SpAG 2 SpAGC 2 SpAGG 3 EATC1B02 F: TGTGGCCCCACTTACTAATATCAG R: CGGGCATTGGTTTACAAGAGTTGC F: ATGGGCTCAAGGATGAATG R: AACTCCAAACGATTGATTTCC F: GTCAACCAACTT F: GTCAACCAACTTGTAAAGCCA R: ACTTGTTTGGCATTTTCCC F: GCTCTTCACGTGTACTTGATC R: TTCGAAGATCCTCCAAGATAC F: TACCATTCAACGCAAGGG R: GTGTATGGTTTTCTTTTCGCA F: CTCCAACATTCCCATGTAGC R: AGCATGTTGTCCCATATAGACC F: TGGCATGAGATTTATGTGGTT R: GTGTGCCACTCAACCTCAC WS00716.F13 F: TCAAGTAATGGACAAACGATACA R: TTTCCAATAGAATGGTGGATTT WS0022.B15 WS0073.H08 WS00111.K13 WS0023.B03 F: TTTGTAGGTGCTGCAGAGATG R: TGGCTTTTTATTCCAGCAAGA F: TGCTCTCTTATTCGGGCTTC R: AAGAACAAGGCTTCCCAATG F: GACTGAAGATGCCGATATGC R: GGCCATATCATCTCAAAATAAAGAA F: AGCAGCTGGGGTCAAAGTT R: AAAGAAAGCATGCATATGACTCAG Velikost PCR produktů (bp) Protokoly PCR: Multiplex 1 (SSR lokusy, koncentrace jejich primerů a příklad fluorescenčního označení forward primerů): PAAC19 F 2 µm (6FAM), PAAC19 R 2 µm SPAGD 1 F 2 µm (PET), SPAGD 1 R 2 µm PAAC23 F 4 µm (6FAM), PAAC23 R 4 µm EATC1B02 F 4 µm (PET), EATC1B02 R 4 µm Forward (F) i revers (R) primery naředíme na zásobní roztoky 100 µm koncentrace (100 pmol/µl) pomocí TE pufru (1 mm Tris HCl, ph 8.0, 0,01 mm EDTA). Při- 12
14 pravíme směs z primerů v požadované koncentraci a v objemu dle počtu analyzovaných vzorků (např. pro objem 100 µl napipetujeme ze zásobního roztoku primerů PAAC19 F 2 µl, PAAC19 R 2 µl, SPAGD 1 F 2 µl, SPAGD 1 R 2 µl, PAAC23 F 4 µl, PAAC23 R 4 µl, EATC1B02 F 4 µl, EATC1B02 R 4 µl a doplníme 76 µl TE pufru). Příprava TE pufru (1 mm Tris HCl, ph 8.0, 0,01 mm EDTA): 10 ml roztoku připravíme z 10 µl 1M Tris HCl a 0,2 µl 0,5M EDTA, doplníme H 2 O (molecular biology reagent) (Sigma Aldrich) do 10 ml. Optimalizovaný protokol PCR s polymerázou Platinum Taq DNA Polymerase (Invitrogen by Life Technologies) s reakční směsí v celkovém objemu 15 µl na 1 vzorek Reakční směs Objem na 1 vzorek 10 x PCR Buffer, minus Mg 1,5 µl 50 mm MgCl 2 0,6 µl 10 mm dntps (2,5 mm each) 0,3 µl Primers mix 0,75 µl Polymerase Platinum Taq 0,075 µl H 2 O (molecular biology reagent) (Sigma Aldrich) 10,775 µl Přidat 1 µl templátové DNA (reagencie Polymerase Platinum Taq, 10 PCR Buffer minus Mg, 50 mm MgCl 2, jsou dodány výrobcem společně s polymerázou) Teplotní režim PCR Krok Teplota Čas Popis ºC 3 min Počáteční denaturace 37 x opakovat od 2. do 4. kroku ºC 45 sec Denaturace ºC 45 sec Annealing ºC 45 sec Elongace 1x ºC 20 min Finální elongace 6. 4 ºC Úložná teplota Chlazení amplifikátů 13
15 Multiplex 2 (SSR lokusy a koncentrace jejich primerů a příklad fluorescenčního označení forward primerů): SpAGC 2 F 2 µm (VIC), SpAGC 2 R 2 µm SpAGG 3 F 2 µm (6FAM), SpAGG 3 R 2 µm Provedeme naředění primerů dle příkladu multiplexu 1. Optimalizovaný protokol PCR s polymerázou Platinum Taq DNA Polymerase (Invitrogen by Life Technologies) s reakční směsí v celkovém objemu 15 µl na 1 vzorek Reakční směs Objem na 1 vzorek 10 x PCR Buffer, minus Mg 1,5 µl 50 mm MgCl 2 0,6 µl 10 mm dntps (2,5 mm each) 0,1 µl Primers mix 0,75 µl Polymerase Platinum Taq 0,075 µl H 2 O (molecular biology reagent) (Sigma Aldrich) 10,775 µl Přidat 1 µl templátové DNA (reagencie Polymerase Platinum Taq, 10 PCR Buffer minus Mg, 50 mm MgCl 2, jsou dodány výrobcem společně s polymerázou) Teplotní režim PCR Krok Teplota Čas Popis ºC 5 min Počáteční denaturace 35 x opakovat od 2. do 4. kroku ºC 45 sec Denaturace ºC 40 sec Annealing ºC 45 sec Elongace 1x ºC 15 min Finální elongace 6. 4 ºC Úložná teplota Chlazení amplifikátů 14
16 Multiplex 3 (SSR lokusy a koncentrace jejich primerů a příklad fluorescenčního označení forward primerů): WS00716.F13 F 2 µm (VIC), WS00716.F13 R 2 µm WS0022.B15 F 2 µm (NED), WS0022.B15 R 2 µm Provedeme naředění primerů dle příkladu multiplexu 1. Optimalizovaný protokol v celkovém objemu 15 µl na 1 vzorek s využitím Type-it Microsatellite PCR Kit (Qiagen, Hilden, Germany) Reakční směs Objem na 1 vzorek H 2 O RNase free 5 µl Qiagen Multiplex 7,5 µl Primers Mix 1,5 µl Přidat 1 µl templátové DNA Teplotní režim PCR Krok Teplota Čas Popis ºC 15 min Počáteční denaturace 26 x opakovat od 2. do 4. kroku ºC 30 sec Denaturace ºC 90 sec Annealing ºC 30 sec Elongace 1x ºC 30 min Finální elongace 6. 4 ºC Úložná teplota Chlazení amplifikátů Uvedené chemikálie kromě primerů jsou součásti komerčního kitu Type-it Microsatellite PCR Kit. 15
17 Multiplex 4 (SSR lokusy a koncentrace jejich primerů a příklad fluorescenčního označení forward primerů): WS0073.H08 F 2 µm (6FAM), WS0073.H08 R 2 µm WS00111.K13 F 2 µm (VIC), WS00111.K13 R 2 µm WS0023.B03 F 2 µm (NED), WS0023.B03 R 2 µm SPAG 2 F 1 µm (NED), SPAG 2 R 1 µm Provedeme naředění primerů dle příkladu multiplexu 1. Optimalizovaný protokol v celkovém objemu 15 µl na 1 vzorek s využitím Type-it Microsatellite PCR Kit (Qiagen, Hilden, Germany) Reakční směs Objem na 1 vzorek H 2 O RNase free 5 µl Qiagen Multiplex 7,5 µl Primers Mix 1,5 µl Přidat 1 µl templátové DNA Teplotní režim PCR Krok Teplota Čas Popis ºC 15 min Počáteční denaturace 26 x opakovat od 2. do 4. kroku ºC 30 sec Denaturace ºC 90 sec Annealing ºC 30 sec Elongace 1x ºC 30 min Finální elongace 6. 4 ºC Úložná teplota Chlazení amplifikátů Uvedené chemikálie kromě primerů jsou součásti komerčního kitu Type-it Microsatellite PCR Kit. 16
18 Požadované přístrojové a materiálové vybavení: vortex, sada pipet a příslušné (sterilní) špičky, mikrozkumavky (stripy, destičky PCR 0,2 ml s víčkem), box chladící na PCR mikrozkumavky, chladící destička, centrifuga, teplotní cyklovač c Postupy elektroforézy v agarózovém gelu Předběžné hodnocení získaných amplifikačních produktů se provádí pomocí horizontální elektroforézy na 2% agarózových gelech. Agaróza (Agarose SERVA Electrophoresis GmbH, Heidelberg) se rozpouští zahříváním v 0,5 TBE pufru (Tris borate EDTA pufr, Duchefa Biochemie B.V.) do získání čirého roztoku. K rozpouštění je vhodné použít mikrovlnnou troubu a proces rozpouštění je nutné sledovat, aby nedošlo k překypění roztoku. K vizualizaci amplifikovaných fragmentů DNA se používá fluorescenční barvivo např. GelRed (GelRed TM Nucleic acid Gel Stain, 10,000XinWater, Biotium, Hayward). GelRed se přidává do zahřátého agarózového roztoku v poměru 1 : Po ztuhnutí gelu se přilije do vany pro elektroforézu 0,5 TBE pufr tak, aby přesahoval asi 0,5 cm nad gel. Do slotů gelu se nanáší PCR amplifikáty (15 µl) smíchané s 4 µl pufru (gel loading buffer, Sigma Aldrich). Pro porovnání velikostí získaných amplifikátů se do vybraného slotu nanese směs: 1 µl standardu 100 bp DNA ladder (NEW ENGLAND Biolabs), 4 µl destilované vody a 2 µl pufru (gel loading buffer). V elektrickém poli se pohybují záporné fragmenty DNA ke kladné elektrodě, jejich migrační schopnost závisí na jejich relativní hmotnosti (velikosti amplifikátu). Potřebná doba trvání elektroforézy je kolem 30 minut při napětí 60 V a dalších minut při napětí 100 V. Po proběhlé elektroforéze se gely dokumentují pod UV zářením pomocí kamerového systému. DNA fragmenty se v gelovém nosiči projevují jako fluoreskující proužky. Požadované přístrojové a materiálové vybavení: analytické váhy, sada pipet a příslušné (sterilní) špičky, mikrovlnná trouba, vortex, horizontální elektroforéza se zdrojem, dokumentační systém s UV transluminátorem, temnou komorou (Darkroom), snímací kamerou a softwarem pro zobrazení gelů 17
19 d Postupy fragmentační analýzy a hodnocení PCR produktů Přesné zjištění velikostí amplifikovaných fragmentů v hodnotách párů bází se provádí na genetickém analyzátoru (např. typu Applied Biosystem 3500). Polymerázová řetězová reakce musí být provedena s fluorescenčně označenými primery (pro uvedený typ analyzátoru na 5 konci s modifikacemi 6FAM, VIC, NED, PET). PCR amplifikáty po 1 µl se nanáší do 96 jamkových destiček pro sekvenátory a před fragmentační analýzou jsou denaturovány. Ke každému vzorku se přidá krátce promíchaná (pomocí vortexu) směs Formamidu (Hi-Di TM Formamide, Applied Biosystem) o objemu 11 µl na jeden vzorek a velikostního standardu (Gene Scan TM 600 LIZ Size standard v 2.0, Applied Biosystem) 0,4 µl na jeden vzorek, destička se krátce stočí na centrifuze a provede se inkubace 4 minuty při teplotě 94 C, pak následuje prudké zchlazení na ledu po dobu minimálně 2 minut. Genetický analyzátor pracuje na principu elektroforetického rozdělení fragmentů DNA v tenké kapiláře naplněné speciálním polymerem. Polymer i zkoumaný vzorek je do kapiláry naplňován automaticky. Detekce fragmentovaných úseků je založena na hodnocení fluorescence z fluorescenčně označených primerů po excitaci laserovým zářením. Přístroj je schopen souběžně detekovat vícebarevnou fluorescenci, to umožňuje v multiplexovém uspořádání hodnotit najednou více markerů, což je z časových i finančních důvodů velmi výhodné. Pro jejich správnou identifikaci po proběhlé analýze je nutné jejich kombinaci sestavit z hlediska velikosti jejich alel a fluorescenčního zabarvení primerů. Dvanáct vybraných lokusů lze seskupit pro fragmentační analýzu do 2 běhů. Pro první běh se skupina skládá z PCR produktů multiplexů 1 a 3 (amplifikované lokusy PAAC19, PAAC23, SpAGD 1, EATC1B02, WS00716.F13, WS0022.B15) jednotlivých rostlinných vzorků, kdy se od každého multiplexu napipetuje po 1 µl, objem směsi formamidu a velikostního standardu se sníží na 10 µl, aby se pro jamku zachoval celkový objem 12 µl, a provede se popsaným způsobem denaturace. V druhém běhu probíhá zároveň fragmentační analýza PCR produktů 2. a 4. multiplexu (amplifikované lokusy WS0073.H08, WS00111.K13, WS0023.B03, SpAG 2, SpAGG 3, SpAGC 2 ) a příprava probíhá stejně jako pro vzorky u prvního běhu. Hodnocení velikosti amplifikačních produktů se provádí pomocí softwarového programu GeneMapper 4.1 (Applied Biosystems), který z výsledku měření velikostního standardu, jenž je přidáván ke každému vzorku, stanoví kalibrační křivku a na jejím podkladě ohodnotí velikosti analyzovaných fragmentů. 18
20 Požadované přístrojové a materiálové vybavení: sada pipet a příslušné špičky, vortex, teplotní cyklovač, centrifuga, genetický analyzátor e Zpracování molekulárních dat Smrk ztepilý má diploidní sadu chromozomů. U sledovaného lokusu získáme pro každého jedince dvě shodné alely v případě homozygota nebo v případě heterozygota dvě různé hodnoty alel. Pro získání genetických charakteristik se velikosti alel z hodnocených lokusů pro sledované populace statisticky zpracovávají (např. za využití statistických programů GenAlEx 6.5 (Peakall, Smouse 2006, 2012), CER- VUS (Kalinowski et al. 2007), GENEPOP 4.2 (Raymond, Rousset 1995; Rousset 2008), STRUCTURE (Pritchard et al. 2000; Falush et al. 2003, 2007; Hubisz et al. 2009). Pro kontrolu velikostí odečtených hodnot mikrosatelitových lokusů včetně ohodnocení frekvence nulových alel lze použit software Micro-Checker (Van Oosterhout et al. 2004). Na základě získaných genetických charakteristik lze u populací hodnotit a porovnávat úroveň genetické diverzity, alelické varianty a frekvence alel, genetické diferenciace mezi populacemi, očekávané a pozorované heterozygotnosti, odchylky od Hardy-Weinbergovy rovnováhy, odvození populačních struktur apod. Jedna z nejvýznamnějších genetických charakteristik je ohodnocení genetických vzdáleností mezi populacemi, které lze vypočítat na základě Neiovy standardní genetické vzdálenosti (Nei 1972). V případě ověřování klonové identity se porovnávají hodnoty alel sledovaných lokusů u jedinců (ramet) příslušných klonů. Výsledkem je zpracovaná genotypizace přehled hodnot alel zvolených lokusů pro šetřené klony. Příslušnost jedince ke klonu je deklarovaná, když jsou shodné hodnoty alel u všech analyzovaných lokusů. V případě použití vysoce polymorfních markerů lze počet sledovaných markerů snížit a tím snížit i náklady na analýzy. Někteří autoři (např. Schueler et al. 2003; Lacis et al. 2009, 2011) uvádějí ve svých pracech využití jen tří až sedmi vysoce polymorfních markerů. V případě zde uvedených markerů pro smrk ztepilý je dostačující pro identifikaci totožných jedinců vybrat 7 vysoce polymorfních SSR markerů (PAAC19, SpAGD 1, SpAGC 2, SpAGG 3, WS00716.F13, WS00111.K13, WS0023.B03). 19
21 III SROVNÁNÍ NOVOSTI POSTUPŮ Novost postupů pro ověřování úrovně genetické diverzity vybraných porostů nebo populací smrku ztepilého se nachází ve využití širšího spektra odzkoušených polymorfních jaderných mikrosatelitových markerů (PAAC23, PAAC19, EATC1B02, SpAGD 1, SpAGG 3, SpAGC 2, SpAG 2, WS00716.F13, WS0022.B15, WS0073.H08, WS00111.K13, WS0023.B03) a v případě identifikace deklarovaných klonů ve využití vybraných 7 lokusů (PAAC19, SpAGD 1, SpAGC 2, SpAGG 3, WS00716.F13, WS00111.K13, WS0023.B03) s vysokou mírou polymorfismu pro snížení nákladů analýz. Uvedené metodické postupy pro ověřování klonové identity (genetické totožnosti) roubovanců ve stávajících semenných sadech by měly být také využity při naplňování dotačního titulu pro nově uznávané semenné sady. Výstupem analýz je zpracovaná multilokusová genotypizace, což představuje databázi velikostí alel hodnocených lokusů k příslušným klonům. Objektivní genetické hodnocení spočívá v přímé analýze DNA jedinců s využitím polymorfních jaderných mikrosatelitových lokusů, jejichž velikosti jsou s přesností jednotek bází zaznamenávány na genetickém analyzátoru Applied Biosystems. Metodika byla vyvíjena na vybraných porostech genové základny GZ 102 Trčkov Šerlišský kotel Vrchmezí a byly k nim přidány i autochtonní smrkové porosty Z CHKO Jeseníky a porost původem z Krušných hor. I když se jedná o porosty pouze z území ČR s vysokou mírou toku genů, při výběru 12 vysoce polymorfních lokusů byly zjištěny genetické rozdíly ve strukturování sledovaných porostů na základě analýzy Structure. Genetické hodnocení bylo provedeno zpočátku s 15 mikrosatelitovými markery a 3 jsme z důvodu nižších hodnot polymorfismu z hodnocení u těchto porostů vyloučili (WS0092A.19, EATC1E03, EATC1G02). Osm zařazených markerů (PAAC23, PAAC19, EATC1B02, WS00716.F13, WS0022.B15, WS0073.H08, WS00111.K13, WS0023.B03) bylo vyvinuto z oblasti kódujících sekvencí DNA (expressed sequence tags - EST), což je pro populační genetiku lesních dřevin přínosné i z hlediska získávání poznatků o variabilitě realizovaných vlastností, které mohou např. souviset s informacemi o proměnlivosti adaptivních znaků (Scotti et al. 2000). Pro získání jednoznačných a reprodukovatelných velikostí amplifikovaných lokusů byly optimalizovány postupy polymerázové řetězové reakce (PCR) a optimalizace probíhaly i z hlediska seskupení analyzovaných lokusů do multiplexů, což představuje ekonomické i časové úspory při zpracování velkého počtu vzorků. Amplifikace vybraných 12 lokusů byly sestaveny do čtyř multiplexů a fragmentační analýzy na genetickém analyzátoru do dvou běhů. 20
22 IV POPIS UPLATNĚNÍ METODIKY Metodika byla optimalizována za účelem získání genetických parametrů pro ověřování úrovně genetické diverzity u smrkových porostů a pro ověřování deklarované identity klonů. Znalosti o genetické struktuře porostů představují zásadní informaci pro opatření směřující k zachování biodiverzity lesních ekosystémů. Na základě stanovení úrovně genetické diverzity lze předpovědět další vývoj populací. Získané poznatky o úrovni genetické diverzity, diferenciaci, heterozygotnosti a dalších genetických charakteristik u zdrojových porostů reprodukčního materiálu jsou také velmi důležité z hlediska převažující umělé obnovy lesa. Informace o genetických charakteristikách porostů mohou být využity jako podklady pro rozhodovací řízení, strategické plánování a legislativní činnost státní správy v oblasti ochrany a reprodukce genofondu lesních dřevin a nakládání s reprodukčním materiálem. Další možné využití získaných poznatků při aplikaci předkládané metodiky je při vyhlašování nebo revizi genových základen a dále v rámci uznávání zdrojů reprodukčního materiálu a jejich zařazování do Národního programu ochrany a reprodukce genofondu lesních dřevin včetně zařazování vzorků z těchto zdrojů do Národní banky osiva a explantátů lesních dřevin, kdy lze především na základě míry genetické diverzity rozhodnout, které populace (porosty) je možné zařadit jako cenné zdroje. Poznatky o diverzitě populací také napomáhají plnit cíle Státní politiky životního prostředí a mezinárodní závazky ČR při ochraně biologické rozmanitosti. Další významné uplatnění metodiky na základě polymorfních mikrosatelitových markerů je pro ověřování klonové identity například v semenných sadech, klonových archivech atd. Genetické otestování deklarovaného původu reprodukčního materiálu umožňuje objektivní kontrolou potvrdit nebo vyvrátit klonovou identitu ramet. Metodické postupy ověřování identity klonů/ortetů pro semenné sady (popř. klonové archivy) budou sloužit pro potřeby státní správy a koordinátora Národního programu ochrany a reprodukce genofondu lesních dřevin (ÚHÚL) pro aplikaci nových kontrolních metod a dotační politiky ČR. Metodika popisuje postupy zpracování vzorků, izolace DNA, amplifikace vybraných lokusů, elektroforézy, fragmentační analýzy a zpracování molekulárních dat. Byly zvoleny mikrosatelitové markery s vysokou mírou polymorfismu, které byly odzkoušeny u autochtonních porostů z Orlických hor, Jeseníků a Krušných hor. V příloze jsou uvedeny příklady výstupů genetických charakteristik ukazující na genetické rozdíly šetřených porostů smrku ztepilého. Možnosti uplatnění těchto postupů v dalších laboratořích může komplikovat finanční náročnost přístrojového vybavení pro fragmentační analýzy, zejména genetického analyzátoru. Fragmen- 21
23 tační analýzy si však lze objednat na zakázku u komerčních firem (např. SEQme, Genomac, Amplicon). V EKONOMICKÉ ASPEKTY Významným ekonomickým aspektem uvedených metodických postupů vedoucích k poznatkům o genetické kvalitě především diverzitě a heterozygotnosti populací nebo porostů je přínos celospolečenský. Současně dochází i k naplňování cílů Národního programu zachování a reprodukce genofondu lesních dřevin podporovat kvalitní genetické zdroje a ověřovat jejich identitu metodou DNA markerů. Reprodukce genově bohatších populací zaručuje získání stabilnějších a odolnějších porostů, které budou zvyšovat biologickou rozmanitost, lépe se přizpůsobovat možným změnám klimatu, a tím přispívat k ochraně životního prostředí. Při posuzování ekonomických aspektů nelze opomenout, že ochrana biologické rozmanitosti představuje také naplnění cílů aktualizované Státní politiky životního prostředí České republiky schválené usnesením vlády č. 6 ze dne 9. ledna 2013 a Strategie ochrany biologické rozmanitosti České republiky schválené usnesením vlády ČR č. 193 ze dne 9. března Vedle celospolečenských přínosů se dá předpokládat i zvýšení tržeb z těžby dřeva u vlastníků lesů v podobě budoucích zvýšených výnosů smrkových porostů založených z kvalitního reprodukčního materiálu. Smrk ztepilý je v našich podmínkách z hlediska jeho hospodářského uplatnění nejvíce rozšířený druh dřeviny, který zásadním způsobem ovlivňuje ekonomiku většiny lesních majetků všech forem vlastnictví, a tedy i celého lesního hospodářství a navazujícího dřevozpracujícího průmyslu (celého lesnicko-dřevařského sektoru). Při reprodukci porostů z kvalitních genetických zdrojů je vyšší záruka, že bude v době mýtní zralosti lesních porostů dosaženo současně i vyšší objemové produkce. Při rozdílu porostních zásob vztažených k obmýtí u kvalitnějších smrkových porostů nacházejících se v současných genových základnách (GZ) oproti ostatním smrkovým porostům na území ČR v objemu 81,04 m 3 /ha a při průměrné ceně surového dříví za rok 2014 ve výši Kč/m 3 činí zvýšené tržby z prodeje dřeva u kvalitnějších smrkových porostů (v GZ) částku Kč/ha. Kalkulace byla provedena na základě těchto informačních zdrojů: 22
24 Analýza datového skladu ÚHÚL DS ERMA (Pařízková, Hradec Králové, 2014) Průměrná relativní bonita dřevin v ČR (Zpráva o stavu lesa a lesního hospodářství 1995, str. 96) Černý, M., Pařez, J. - Malík, Z: Růstové a taxační tabulky hlavních dřevin České republiky (smrk, borovice, buk, dub), IFER, 1996 Vyhlášení průměrné ceny dřeva pro rok 2015 k výpočtu poplatku za odnětí lesních pozemků surového dříví (Věstník MZe, částka 2, str. 73, prosinec 2014) Vzhledem ke skutečnosti, že průměrná cena surového dříví v ČR, která je každoročně zveřejňována ve Věstníku MZe, v roce 2014 činila Kč/m 3 a reprezentuje cenu dřeva na pni, tj. cenu surového dříví na odvozním místě po odečtení těžebních nákladů, je možné tento ekonomický efekt současně označit za zvýšený čistý výnos získaný ze smýceného lesního porostu v době obmýtí, který se projeví i ve zvýšeném hospodářském výsledku (zisku) vlastníků lesa. Ve skutečnosti lze očekávat ekonomický přínos pro vlastníka lesa ještě vyšší, pokud porovnáme obě hodnoty mýtní výtěže v obmýtí (v GZ oproti ostatním smrkovým porostům) vypočtené na základě obvykle dosahovaného procentického podílu jednotlivých sortimentů surového dříví a jejich cen (především pilařských výřezů) a obvyklých těžebních nákladů v místě a čase. Vyšší celková objemová produkce porostů z kvalitních genetických zdrojů se však neprojeví v ekonomice vlastníků lesa pouze v době obmýtí, ale již také v rámci výchovných opatření v podobě zvýšených příjmů z provedených probírek (z předmýtních těžeb), což rovněž pozitivně ovlivní ekonomickou situaci vlastníků lesa a přispěje k podpoře ekonomického pilíře trvale udržitelného hospodaření v lese. Vzhledem k současné kalamitní situaci u smrkových porostů je zhodnocení ekonomického přínosu vztaženo k období předcházejících hospodářských výsledků. Náklady na postupy genetických analýz uvedených v metodice jsou kalkulovány na spotřební materiál a chemikálie s předpokladem přístrojového laboratorního vybavení pro analýzy DNA. Na izolaci DNA vzorku z jednoho stromu jsou průměrné náklady 109 Kč. Náklady u jednoho vzorku (stromu) na PCR produkty a následné fragmentační analýzy činí pro 12 lokusů 155 Kč vč. DPH, za předpokladu provedení analýz v multiplexech dle uvedených optimalizovaných postupů. V uvedené kalkulaci našich nákladů (výzkumné pracoviště) nejsou zahrnuty doplňkové náklady, náklady na odpisy přístrojového vybavení, osobní náklady, náklady na vývoj metod, které jsou odvislé od konkrétní situace vybavenosti (materiální i perso- 23
25 nální) pracoviště. Při využití služeb komerčních laboratoří jsme při průzkumu na tuzemském trhu zjistili výhodné cenové nabídky od firmy SEQme. Předpokládaná cena na analýzu jednoho vzorku je 242 Kč včetně DPH a zahrnuje PCR amplifikaci a fragmentační analýzu jednoho multiplexu. Tato cena je kalkulována při vlastním dodání vysoce kvalitní DNA a vypracovaných metodických postupů (nebude nutno dopracovávat jakékoliv optimalizace PCR). Na postupy optimalizace PCR komerční firmy nevedou ceníky, např. firma SEQme si tyto práce účtuje hodinovou sazbou a to Kč bez DPH/hod. Fragmentační analýza při dodání vlastních PCR amplifikátů v destičce vychází na 72 Kč pro jeden vzorek (z 1 stromu 1 multiplex). VI DEDIKACE Metodika je výsledkem řešení výzkumného projektu NAZV č. QJ (Modelový postup molekulárně genetické charakterizace genové základny jako podklad pro účely rozhodování státní správy v oblasti zachování a reprodukce genetických zdrojů). 24
26 VII SEZNAM POUŽITÉ SOUVISEJÍCÍ LITERATURY Bozhko M., Riegel R., Schubert R., Müller-Starck G A cyclophilin gene marker confirming geographical differentiation of Norway spruce populations and indicating viability response on excess soil-born salinity. Molecular Ecology, 12: Cvjetković B., Konnert M., Fussi B., Mataruga M., Šijačić-Nikolić M., Daničić V., Lučić A Norway spruce (Picea abies Karst.) variability in progeny tests in Bosnia and Herzegovina. Genetika, 49: Falush D., Stephens M., Pritchard J.K Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies. Genetics, 164 (4): Falush D., Stephens M., Pritchard J.K Inference of population structure using multilocus genotype data: dominant markers and null alleles. Molecular Ecology, 7 (4): Geburek T Genetic variation of Norway spruce (Picea abies [L.] Karst.) populations in Austria. III. Macrospatial allozyme patterns of high elevation populations. Forest Genetics, 6: Hampe A., Petit R.J Conserving biodiversity under climate change: The rear edge matters. Ecology Letters, 8: Hubisz M.J., Falush D., Stephens M., Pritchard J.K Inferring weak population structure with the assistance of sample group information. Molecular Ecology Resources, 9 (5): Kalinowski S.T., Taper M.L., Marshall T.C Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 16: Konnert M Genetic variation of Picea abies in southern Germany as determined using isozyme and STS markers. Dendrobiology, 61: Lacis G., Rashal I., Ruisa S., Trajkovski V., Iezzoni A.F Assessment of genetic diversity of Latvian and Swedish sweet cherry (Prunus avium L.) genetic resources collection by using SSR (microsatellite) markers. Scientia Horticulturae, 121:
27 Lacis G., Rashal I., Trajkovski V Implementation of a limited set of SSR markers for screening of genetic variability in Latvian and Swedish sour cherry (Prunus cerasus L.) genetic resources collections. Proceedings of the Latvian Academy of Sciences, 65 (1/2) (672/673): DOI: /v Maghuly F., Pinsker W., Praznik W., Fluch S Genetic diversity in managed subpopulations of Norway spruce [Picea abies (L). Karst.]. Forest Ecology and Management, 222: Maghuly F., Burg K., Pinsker W., Nittinger F., Praznik W., Fluch S Short Note: Development of mitochondrial markers for population genetics of Norway Spruce [Picea abies (L). Karst]. Silvae Genetica, 57: Melnikova M.N., Petrov N.B., Lomov A.A., la Porta N., Politov D.V Testing of Microsatellite Primers with Different populations of Eurasian Spruces Picea abies (L.) Karst. and Picea obovata Ledeb. Russian Journal of Genetics, 48: Meloni M., Perini D., Binelli G The distribution of genetic variation in Norway spruce (Picea abies [L.] Karst.) populations in the western Alps. Journal of Biogeography, 34: Neale D.B., Kremer A Forest tree genomics: Growing resources and applications. Nature Reviews Genetics, 12: Nei M Genetic distance between populations. American Naturalist, 106: Nowakowska J.A., Zachara T., Konecka A Genetic variability of Scots pine (Pinus sylvestris L.) and Norway spruce (Picea abies L. Karst.) natural regeneration compared with their maternal stands. Leśne Prace Badawcze, 75 (1): Peakall R., Smouse P.E GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 6: Peakall R., Smouse P.E GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research an update. Bioinformatics, 28: Pfeiffer A.M., Oliviery A.M., Morgante M Identification and characterization of Microsatellites in Norway spruce (Picea abies K.). Genome, 40:
28 Pritchard J.K., Stephens M., Donnelly P Inference of population structure using multilocus genotype data. Genetics, 155 (2): Raymond M., Rousset F GENEPOP (version 1.2): population genetics software for exact tests and ecumenicism. Journal of Heredity, 86: Rousset F Genepop 007: a complete reimplementation of the Genepop software for Windows and Linux. Molecular Ecology Resources, 8: Rungis D., Bérubé Y., Zhang J., Ralph S., Ritland C.E., Ellis B.E., Douglas C., Bohlmann J., Ritland K Robust simple sequence repeat markers for spruce (Picea spp.) from expressed sequence tags. Theoretical and Applied Genetics, 109: Scotti I., Magni F., Fink R., Powell W., Binelli G., Hedley P.E Microsatellite repeats are not randomly distributed within Norway spruce (Picea abies K.) expressed sequences. Genome, 43: Scotti I., Magni F., Paglia G. P., Morgante M Trinucleotide microsatellites in Norway spruce (Picea abies): their features and the development of molecular markers. Theoretical and Applied Genetics, 106: Scotti I., Pagila G., Magni F., Morgante M Population genetics of Norway spruce (Picea abies Karst.) at regional scale: sensitivity of different microsatellite motif classes in detecting differentiation. Annals of Forest Science, 63: Schubert R., Mueller-Starck G., Riegel R Development of EST-PCR markers and monitoring their intrapopulational genetic variation in Picea abies (L.) Karst. Theoretical and Applied Genetics, 103: Schueler S., Tusch A., Schuster M., Ziegenhagen B Characterization of microsatellites in wild and sweet cherry (Prunus avium L.) markers for individual identification and reproductive processes. Genome, 46: Tollefsrud M.M., Sønstebø J.H., Brochmann C., Johnsen Ø., Skroppa T., Vendramin G.G Combined analysis of nuclear and mitochondrial markers provide new insight into the genetic structure of North European Picea abies. Heredity, 102: Svoboda P Lesní dřeviny a jejich porosty. Část I. Státní zemědělské nakladatelství, Praha. Unger G. M., Konrad H., Geburek T Does spatial genetic structure increase with altitude? An answer from Picea abies in Tyrol, Austria. Plant Systematics and Evolution, 292:
29 Úradníček L., Maděra P., Tichá S., Koblížek J Dřeviny České republiky. Kostelec nad Černými lesy, Lesnická práce: 367 s. Van Oosterhout C., Hutchinson W.F., Wills D.P.M., Shipley P Micro-Checker: software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4: Vendramin G.G., Anzidei M., Madaghiele A., Sperisen C., Bucci G Chloroplast microsatellite analysis reveals the presence of population subdivision in Norway spruce (Picea abies K.). Genome, 43: Verbylaitè R., Pliūra A., Lygis V., Suchockas V., Jankauskienė J., Labokas J Genetic diversity and its spatial distribution in self-regenerating Norway spruce and Scots pine stands. Forests, 8: 470; doi: /f Whitham T.G., Bailey J.K., Schweitzer J.A., Shuster S.M., Bangert R.K., LeRoy C.J., Lonsdorf E.V., Allan G.J., DiFazio S.P., Potts B.M. et al A framework for community and ecosystem genetics from genes to ecosystems. Nature Reviews Genetics, 7:
30 VIII SEZNAM PUBLIKACÍ, KTERÉ PŘEDCHÁZELY METODICE Cvrčková H., Máchová P., Dostál J., Malá J.: Sledování genetické proměnlivosti chlumního ekotypu smrku ztepilého pomocí RAPD. (Studying of genetic variability of Norway spruce hurst ecotype by RAPD). Zprávy lesnického výzkumu, 56, 2011(2): Cvrčková H., Máchová P., Dostál J., Malá J.: Hodnocení genetické diversity vybraných populací smrku ztepilého pomocí mikrosatelitových markerů. (Assessment of genetic diversity of selected populations of Norway spruce by microsatellite markers). Zprávy lesnického výzkumu, 58, 2013 (3): Máchová P., Cvrčková H., Malá J. Využití mikrosatelitových markerů pro hodnocení semenného sadu smrku ztepilého (Evaluation of Norway spruce seed orchard using microsatellite markers). Zprávy lesnického výzkumu, 59, 2014 (4): Cvrčková H., Máchová P. Genetická charakterizace smrku ztepilého pomocí mikrosatelitových markerů. Lesnický průvodce, 2015, 8: 36 s. Máchová P., Trčková O., Cvrčková H. Use of Nuclear Microsatellite Loci for Evaluating Genetic Diversity of Selected Populations of Picea abies (L.) Karsten in the Czech Republic. Forests, 9, 2018: 92; doi: /f Cvrčková H., Máchová P., Trčková O. Genetická variabilita vybraných porostů smrku ztepilého z Jeseníků, Orlických a Krušných hor. Zprávy lesnického výzkumu. Zprávy lesnického výzkumu, 63, 2018 (4): in press. 29
31 USE OF MICROSATELLITE MARKERS FOR EVALUATING GENETIC DIVERSITY OF NORWAY SPRUCE Summary Norway spruce (Picea abies (L.) Karsten) is a coniferous species belonging to the family Pinaceae, which is the most economically important tree species in Central and Northern Europe. This species is very demanding of soil moisture and requires higher relative humidity. Today s significant reduction in the occurrence of spruce in some localities is due to the natural increase in drought caused by climate change and by infestation with eight-toothed spruce bark beetle. It is widely assumed that populations characterized by narrow genetic diversity could be more sensitive to environmental changes or diseases, thereby leading to a decrease in productivity. Therefore, it is essential to determine the genetic variability in economically and ecologically valuable stands and populations. This methodology presents the use of DNA analyses by nuclear microsatellite markers for genetic characterization and clonal identification of Picea abies (L.) Karsten. Microsatellites, also known as simple sequence repeats (SSR) are small repetitive DNA sequences that are highly variable markers and commonly used in population genetic. The selected fourteen Norway spruce stands (from Jeseníky, Orlické hory, Krušné hory) were used to develop this methodology. This methodology describes the processes of sampling, isolation of DNA, conditions of polymerase chain reaction (PCR), separation, sizing of amplification products and calculations of molecular data. Total genomic DNA was extracted using a DNeasy Plant Mini Kit (QIAGEN, Venlo, Netherlands) from 100 mg fresh needles or 20 mg lyophilized needles. The SSR method is based on the polymerase chain reaction (PCR) with specific primers. Twelve variable microsatellite markers PAAC23, PAAC19 (Scotti et al. 2000), EATC1B02 (Scotti et al. 2002), SpAGD 1, SpAGG 3, SpAGC 2, SpAG 2 (Melnikova et al. 2012), WS00716.F13, WS0022.B15, WS0073.H08, WS00111.K13, WS0023.B03 (Rungis et al. 2004) were selected for DNA analyzes. PCR products were separated by capillary electrophoresis using the Applied Biosystems 3500 genetic analyser. The identified loci were verified as highly polymorphic and could be used as markers for the next genetic analyses of populations and for verifying the clonal identity of the Norway spruce. Acquiring new knowledge on the genetic structure of coniferous species populations, especially in relation to valuable ecotypes of Norway spruce, is very important in 30
32 order to maintain the ecological stability of forests and to optimize the timber production. The developed procedures of genetic monitoring with DNA markers will be used in the amendment of forestry legislation and in the state subsidy policy in the area of protection and reproduction of forest tree gene resources. Knowledge based on DNA analyses regarding the variability of genetic resources will contribute to the quality of the reproduction material and the creation of species composition in forests. 31
33 32
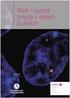
34 Příloha Příklady výstupů genetických analýz šetřených porostů smrku ztepilého Obr. 1: Ukázka výstupu fragmentační analýzy z genetického analyzátoru (záznam velikostí alel) 33
35 Tab. 2: Průměrné hodnoty genetických charakteristik pro sledované porosty smrku ztepilého Porosty N Na Ne I Priv. alely H o H e F SM KH 24 13,17 8,67 2,23 0,67 0,81 0,85 0,031 SM S ,75 9,10 2,27 0,33 0,77 0,85 0,077 SM S ,25 9,48 2,29 0,25 0,76 0,85 0,087 SM T ,92 9,82 2,36 0,42 0,75 0,86 0,107 SM T ,75 9,51 2,28 0,33 0,75 0,84 0,085 SM V ,67 8,69 2,21 0,25 0,74 0,84 0,101 SM V ,67 8,60 2,25 0,33 0,77 0,85 0,072 SM J ,08 9,23 2,25 0 0,70 0,85 0,170 SM J ,92 8,25 2,21 0,33 0,73 0,84 0,093 SM J ,92 8,80 2,16 0,08 0,65 0,82 0,195 SM J ,33 8,59 2,24 0,08 0,71 0,84 0,134 SM J ,25 7,68 2,11 0 0,67 0,83 0,164 SM J ,50 8,57 2,16 0,17 0,68 0,81 0,141 SM J ,42 8,19 2,19 0,17 0,66 0,83 0,190 Průměr/Mean 31,6 14,11 8,80 2,23 0,24 0,73 0,84 0,118 N: počet vzorků, Na: počet různých alel, Ne: počet efektivních alel, I: Shannonův informační index, Priv. alely: počet privátních alel, H o : heterozygotnost pozorovaná, H e : heterozygotnost očekávaná, F: fixační index Obr. 2: Výsledky analýzy hlavních koordinát (PCoA) 34
36 Obr. 3: Zhodnocení genetické struktury Bayesianskou metodou pro K = 3 u sledovaných porostů (1 SM KH, 2 SM S1, 3 SM S4, 4 SM T1, 5 SM T4, 6 SM V1, 7 SM V4, 8 SM J03, 9 SM J11, 10 SM J14, 11 SM J25, 12 SM J51, 13 SM J92, 14 SM J94) 35
GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ
 GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE Certifikované METODIKY Certifikovaná metodika Ing. HELENA CVRČKOVÁ, Ph.D Ing. PAVLÍNA MÁCHOVÁ, Ph.D 8/2015 Genetická
GENETICKÁ CHARAKTERIZACE SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE Certifikované METODIKY Certifikovaná metodika Ing. HELENA CVRČKOVÁ, Ph.D Ing. PAVLÍNA MÁCHOVÁ, Ph.D 8/2015 Genetická
LESNICKÝ PRŮVODCE VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ. C er ti f i k ov a n é
 VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. HELENA CVRČKOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO OVĚŘOVÁNÍ KLONOVÉ IDENTITY U TŘEŠNĚ PTAČÍ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Ing. HELENA CVRČKOVÁ, Ph.D. Ing. OLGA TRČKOVÁ
LESNICKÝ PRŮVODCE HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ. C er ti f i k ov a n é
 HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr.
HODNOCENÍ GENETICKÝCH CHARAKTERISTIK U BOROVICE LESNÍ S VYUŽITÍM MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr.
IZOLACE DNA (KIT DNeasy Plant Mini)
 17.1 Izolace DNA (kit DNeasy Plant Mini) Strana 1 IZOLACE DNA (KIT DNeasy Plant Mini) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku pomocí komerčního kitu DNeasy Plant
17.1 Izolace DNA (kit DNeasy Plant Mini) Strana 1 IZOLACE DNA (KIT DNeasy Plant Mini) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku pomocí komerčního kitu DNeasy Plant
LESNICKÝ PRŮVODCE GENETICKÁ CHARAKTERIZACE JEDLE BĚLOKORÉ POMOCÍ MIKROSATELITOVÝCH MARKERŮ. C er ti f i k ov a n é
 GENETICKÁ CHARAKTERIZACE JEDLE BĚLOKORÉ POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. 5/2016 Genetická charakterizace
GENETICKÁ CHARAKTERIZACE JEDLE BĚLOKORÉ POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. 5/2016 Genetická charakterizace
VYUŽITÍ MIKROSATELITOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZTEPILÉHO
 VYUŽIÍ MIKROSAELIOVÝCH ZPRÁVY LESNICKÉHO MARKERŮ PRO VÝZKUMU, HODNOCENÍ 59, 2014 SEMENNÉHO (4): 243-249 SADU SMRKU ZEPILÉHO VYUŽIÍ MIKROSAELIOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZEPILÉHO EVALUAION
VYUŽIÍ MIKROSAELIOVÝCH ZPRÁVY LESNICKÉHO MARKERŮ PRO VÝZKUMU, HODNOCENÍ 59, 2014 SEMENNÉHO (4): 243-249 SADU SMRKU ZEPILÉHO VYUŽIÍ MIKROSAELIOVÝCH MARKERŮ PRO HODNOCENÍ SEMENNÉHO SADU SMRKU ZEPILÉHO EVALUAION
Jednotné pracovní postupy zkoušení krmiv
 Národní referenční laboratoř Strana KVANTITATIVNÍ STANOVENÍ GENETICKÝCH MODIFIKACÍ METODOU qpcr POMOCÍ ROTOR-GENE PROBE PCR KITU Účel a rozsah Postup slouží ke kvantitativnímu stanovení genetických modifikací
Národní referenční laboratoř Strana KVANTITATIVNÍ STANOVENÍ GENETICKÝCH MODIFIKACÍ METODOU qpcr POMOCÍ ROTOR-GENE PROBE PCR KITU Účel a rozsah Postup slouží ke kvantitativnímu stanovení genetických modifikací
STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE
 STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr. LUCIE POLÁKOVÁ
STUDIUM VARIABILITY POPULACÍ BUKU LESNÍHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ LESNICKÝ PRŮVODCE C er ti f i k ov a n é METODIKY Ing. HELENA CVRČKOVÁ, Ph.D. Ing. PAVLÍNA MÁCHOVÁ, Ph.D. Mgr. LUCIE POLÁKOVÁ
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
HODNOCENÍ GENETICKÉ DIVERZITY VYBRANÝCH POPULACÍ SMRKU ZTEPILÉHO POMOCÍ MIKROSATELITOVÝCH MARKERŮ
 HODNOCENÍ GENEICKÉ DIVERZIY ZPRÁVY VYBRANÝCH LESNICKÉHO POPULACÍ VÝZKUMU, SMRKU 58, ZEPILÉHO 2013 (3): POMOCÍ 273-279 MIKROSAELIOVÝCH MARKERŮ HODNOCENÍ GENEICKÉ DIVERZIY VYBRANÝCH POPULACÍ SMRKU ZEPILÉHO
HODNOCENÍ GENEICKÉ DIVERZIY ZPRÁVY VYBRANÝCH LESNICKÉHO POPULACÍ VÝZKUMU, SMRKU 58, ZEPILÉHO 2013 (3): POMOCÍ 273-279 MIKROSAELIOVÝCH MARKERŮ HODNOCENÍ GENEICKÉ DIVERZIY VYBRANÝCH POPULACÍ SMRKU ZEPILÉHO
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU Jednou z nejvhodnějších metod pro detekci minimální reziduální
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KARCINOMU PANKREATU Jednou z nejvhodnějších metod pro detekci minimální reziduální
Studium polymorfismu u vybraných populací smrku ztepilého Picea abies (L.)
 Výzkum možností minimalizace obsahů organických škodlivin ve zdrojích pitných vod v Krušných horách Studium polymorfismu u vybraných populací smrku ztepilého Picea abies (L.) Karsten pomocí DNA analýz
Výzkum možností minimalizace obsahů organických škodlivin ve zdrojích pitných vod v Krušných horách Studium polymorfismu u vybraných populací smrku ztepilého Picea abies (L.) Karsten pomocí DNA analýz
Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae).
 Populační studie Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae). American Journal of Botany 87(8): 1128
Populační studie Fisher M. & al. (2000): RAPD variation among and within small and large populations of the rare clonal plant Ranunculus reptans (Ranunculaceae). American Journal of Botany 87(8): 1128
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR Jednou z nejvhodnějších metod pro detekci minimální reziduální choroby
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIUDÁLNÍ CHOROBY MRD EGFR Jednou z nejvhodnějších metod pro detekci minimální reziduální choroby
Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis.
 Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER
 PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER Trojan V., Hanáček P., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER Trojan V., Hanáček P., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C
 α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C Popis stripů: Pracovní postup Izolace DNA Doporučujeme použít následující kit pro izolaci DNA z plné krve nebo jiných typů vzorků: Spin Micro DNA Extraction
α-globin StripAssay Kat. číslo 4-160 10 testů 2-8 C Popis stripů: Pracovní postup Izolace DNA Doporučujeme použít následující kit pro izolaci DNA z plné krve nebo jiných typů vzorků: Spin Micro DNA Extraction
ÚVOD. Klíčová slova: smrk ztepilý, RAPD, somatická embryogeneze, polymorfizmus Key words: Norway spruce, RAPD, somatic embryogenesis, polymorphism
 In this study, we describe the use of DNA analysis by RAPD method (Random Amplified Polymorphic DNA) to identify a variability of Norway spruce hurst ecotype. Total genomic DNA was isolated by DNA Plant
In this study, we describe the use of DNA analysis by RAPD method (Random Amplified Polymorphic DNA) to identify a variability of Norway spruce hurst ecotype. Total genomic DNA was isolated by DNA Plant
Genetická diverzita masného skotu v ČR
 Genetická diverzita masného skotu v ČR Mgr. Jan Říha Výzkumný ústav pro chov skotu, s.r.o. Ing. Irena Vrtková 26. listopadu 2009 Genetická diverzita skotu pojem diverzity Genom skotu 30 chromozomu, genetická
Genetická diverzita masného skotu v ČR Mgr. Jan Říha Výzkumný ústav pro chov skotu, s.r.o. Ing. Irena Vrtková 26. listopadu 2009 Genetická diverzita skotu pojem diverzity Genom skotu 30 chromozomu, genetická
Braf V600E StripAssay
 Braf V600E StripAssay Kat. číslo 5-570 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci čerstvých nebo mražených biopsií použijte soupravy Qiagen QIAmp DNA Mini nebo Micro. Pro izolaci
Braf V600E StripAssay Kat. číslo 5-570 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci čerstvých nebo mražených biopsií použijte soupravy Qiagen QIAmp DNA Mini nebo Micro. Pro izolaci
Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK
 MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
NRAS StripAssay. Kat. číslo 5-610. 20 testů 2-8 C
 NRAS StripAssay Kat. číslo 5-610 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci DNA musí být použita vhodná metoda vzhledem k typu tkáně vzorku. Pro doporučení vhodné metody kontaktujte
NRAS StripAssay Kat. číslo 5-610 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Pro izolaci DNA musí být použita vhodná metoda vzhledem k typu tkáně vzorku. Pro doporučení vhodné metody kontaktujte
UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC
 UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC VYUŽITÍ DNA MIKROSATELITŮ POUŽÍVANÝCH V PANELU NA URČOVÁNÍ
UTILIZATION OF DNA MICROSATELLITES USED IN PARENTITY PANEL IN EVALUATION OF DIVERZITY AND DISTANCES BETWEEN THE BREEDS OF PIGS IN CZECH REPUBLIC VYUŽITÍ DNA MIKROSATELITŮ POUŽÍVANÝCH V PANELU NA URČOVÁNÍ
Tkáňový homogenizátor MagNA Lyser od společnosti Roche
 Izolace RNA Pracovní postup Homogenizace: Pozn. Postup homogenizace platí pouze pro izolaci RNA z nativní tkáně, v případě izolace z buněčné suspenze je tento krok vynechán a začíná se přídavkem homogenizačního
Izolace RNA Pracovní postup Homogenizace: Pozn. Postup homogenizace platí pouze pro izolaci RNA z nativní tkáně, v případě izolace z buněčné suspenze je tento krok vynechán a začíná se přídavkem homogenizačního
EGFR XL StripAssay. Kat. číslo 5-630. 20 testů 2-8 C
 EGFR XL StripAssay Kat. číslo 5-630 20 testů 2-8 C Popis stripu Pracovní postup 1. Izolace DNA Pro izolaci DNA použijte vhodný izolační kit. Doporučené kity jsou následující: Pro izolaci čerstvých nebo
EGFR XL StripAssay Kat. číslo 5-630 20 testů 2-8 C Popis stripu Pracovní postup 1. Izolace DNA Pro izolaci DNA použijte vhodný izolační kit. Doporučené kity jsou následující: Pro izolaci čerstvých nebo
cdna synthesis kit First-Strand cdna Synthesis System Verze 1.2
 cdna synthesis kit First-Strand cdna Synthesis System Verze 1.2 Obsah soupravy a její skladování Tato souprava pro reverzní transkripci obsahuje reagencie potřebné k provedení reverzní transkripce (RT)
cdna synthesis kit First-Strand cdna Synthesis System Verze 1.2 Obsah soupravy a její skladování Tato souprava pro reverzní transkripci obsahuje reagencie potřebné k provedení reverzní transkripce (RT)
CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION
 CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION DIGITÁLNÍ OBRAZOVÁ ANALÝZA ELEKTROFORETICKÝCH GELŮ *** Vyhodnocování získaných elektroforeogramů: Pro vyhodnocování
CLP ANALYSIS OF MOLECULAR MARKERS DIGITAL IMAGE ANALYSIS OF ELECTROPHOEROGRAMS CZECH VERSION DIGITÁLNÍ OBRAZOVÁ ANALÝZA ELEKTROFORETICKÝCH GELŮ *** Vyhodnocování získaných elektroforeogramů: Pro vyhodnocování
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin
 Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace praktických cvičení molekulárně-biologických předmětů o sekvenční úlohy PRACOVNÍ PROTOKOL PRO PŘEDMĚT GENETCKÁ DIVERZITA Vypracováno
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace praktických cvičení molekulárně-biologických předmětů o sekvenční úlohy PRACOVNÍ PROTOKOL PRO PŘEDMĚT GENETCKÁ DIVERZITA Vypracováno
DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH. Michaela Nesvadbová
 DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH Michaela Nesvadbová Význam identifikace živočišných druhů v krmivu a potravinách povinností každého výrobce je řádně a pravdivě označit
DNA TECHNIKY IDENTIFIKACE ŽIVOČIŠNÝCH DRUHŮ V KRMIVU A POTRAVINÁCH Michaela Nesvadbová Význam identifikace živočišných druhů v krmivu a potravinách povinností každého výrobce je řádně a pravdivě označit
ISSR (Inter simple sequence repeat) Jiří Košnar
 ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
Soubor Map: Mapa struktury porostů na 7 TVP v CHKO Orlické hory Vacek S., Vacek Z., Bulušek D., Ulbrichová I.
 Soubor Map: Mapa struktury porostů na 7 TVP v CHKO Orlické hory Vacek S., Vacek Z., Bulušek D., Ulbrichová I. Mapa struktury porostu na TVP 5 v CHKO Orlické hory Vacek S., Vacek Z., Bulušek D., Ulbrichová
Soubor Map: Mapa struktury porostů na 7 TVP v CHKO Orlické hory Vacek S., Vacek Z., Bulušek D., Ulbrichová I. Mapa struktury porostu na TVP 5 v CHKO Orlické hory Vacek S., Vacek Z., Bulušek D., Ulbrichová
VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS
 VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS OVĚŘENÍ VHODNOSTI VYUŽITÍ PANELU MIKROSATELITŮ PRO URČENÍ PARENTITY VYBRANÝCH PLEMEN PSŮ Bryndová
VERIFICATION AVAILABILITY MICROSATELLITES PANEL FOR PARENTAGE IDENTIFICATION OF DIFFERENT CANINE BREEDS OVĚŘENÍ VHODNOSTI VYUŽITÍ PANELU MIKROSATELITŮ PRO URČENÍ PARENTITY VYBRANÝCH PLEMEN PSŮ Bryndová
ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD
 ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD Distinguishing of Wheat Varieties (Tritium aestivum L.) by Method RAPD Zuzana Kohutová, Zuzana Kocourková, Hana Vlastníková, Petr Sedlák
ODLIŠENÍ ODRŮD PŠENICE OBECNÉ TRITICUM AESTIVUM L. METODOU RAPD Distinguishing of Wheat Varieties (Tritium aestivum L.) by Method RAPD Zuzana Kohutová, Zuzana Kocourková, Hana Vlastníková, Petr Sedlák
Využití molekulárních markerů v systematice a populační biologii rostlin. 10. Další metody
 Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
Polymorfismus délky restrikčních fragmentů (RFLP)
 ÚSTAV LÉKAŘSKÉ BIOCHEMIE A LABORATORNÍ DIAGNOSTIKY 1. LF UK Polymorfismus délky restrikčních fragmentů (RFLP) Praktické cvičení z lékařské biochemie Všeobecné lékařství Martin Vejražka 2017/18 Obsah POLYMORFISMUS
ÚSTAV LÉKAŘSKÉ BIOCHEMIE A LABORATORNÍ DIAGNOSTIKY 1. LF UK Polymorfismus délky restrikčních fragmentů (RFLP) Praktické cvičení z lékařské biochemie Všeobecné lékařství Martin Vejražka 2017/18 Obsah POLYMORFISMUS
Konečná zpráva hodnocení různých způsobů přípravy vzorků pro AMPLICOR HPV test firmy Roche
 Konečná zpráva hodnocení různých způsobů přípravy vzorků pro AMPLICOR HPV test firmy Roche Charakteristika testu: Set AMPLICOR HPV vyráběný firmou Roche je určený pro detekci vysoko-rizikových typů lidských
Konečná zpráva hodnocení různých způsobů přípravy vzorků pro AMPLICOR HPV test firmy Roche Charakteristika testu: Set AMPLICOR HPV vyráběný firmou Roche je určený pro detekci vysoko-rizikových typů lidských
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION
 USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
Molekulární metody pro střední školy
 Literatura Coltman, D. W. (). Male reproductive success in a promiscuous mammal: behavioural estimates compared with genetic paternity. Mol Ecol. 8:(7): -20. Fornůsková, A. (2007). Mikrosatelity a jejich
Literatura Coltman, D. W. (). Male reproductive success in a promiscuous mammal: behavioural estimates compared with genetic paternity. Mol Ecol. 8:(7): -20. Fornůsková, A. (2007). Mikrosatelity a jejich
Využití DNA markerů ve studiu fylogeneze rostlin
 Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské. doc. RNDr. Ivan Mazura, CSc.
 Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské praxi doc. RNDr. Ivan Mazura, CSc. Historie forenzní genetiky 1985-1986 Alec Jeffreys a satelitní DNA 1980 Ray
Genetický polymorfismus jako nástroj identifikace osob v kriminalistické a soudnělékařské praxi doc. RNDr. Ivan Mazura, CSc. Historie forenzní genetiky 1985-1986 Alec Jeffreys a satelitní DNA 1980 Ray
Kantor P., Vaněk P.: Komparace produkčního potenciálu douglasky tisolisté... A KYSELÝCH STANOVIŠTÍCH PAHORKATIN
 KOMPARACE PRODUKČNÍHO POTENCIÁLU DOUGLASKY TISOLISTÉ NA ŽIVNÝCH A KYSELÝCH STANOVIŠTÍCH PAHORKATIN COMPARISON OF THE PRODUCTION POTENTIAL OF DOUGLAS FIR ON MESOTROPHIC AND ACIDIC SITES OF UPLANDS PETR
KOMPARACE PRODUKČNÍHO POTENCIÁLU DOUGLASKY TISOLISTÉ NA ŽIVNÝCH A KYSELÝCH STANOVIŠTÍCH PAHORKATIN COMPARISON OF THE PRODUCTION POTENTIAL OF DOUGLAS FIR ON MESOTROPHIC AND ACIDIC SITES OF UPLANDS PETR
Metodika analýzy molekulárních markerů u jilmu, Ulmus L.
 Metodika analýzy molekulárních markerů u jilmu, Ulmus L. Metodika byla vypracovaná jako výstup projektu NAZV QI92A247 Charakterizace genetické struktury autochtonních populací jilmů pomocí DNA analýz,
Metodika analýzy molekulárních markerů u jilmu, Ulmus L. Metodika byla vypracovaná jako výstup projektu NAZV QI92A247 Charakterizace genetické struktury autochtonních populací jilmů pomocí DNA analýz,
Braf 600/601 StripAssay
 Braf 600/601 StripAssay Kat. číslo 5-560 20 testů 2-8 C Popis stripu: Pracovní postup Kit umožňuje detekci 9 mutací v genu BRAF (kodón 600 a 601) Další informace najdete v OMIM Online Mendelian Inheritance
Braf 600/601 StripAssay Kat. číslo 5-560 20 testů 2-8 C Popis stripu: Pracovní postup Kit umožňuje detekci 9 mutací v genu BRAF (kodón 600 a 601) Další informace najdete v OMIM Online Mendelian Inheritance
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Soubor map současného rozšíření lesních dřevin v Krkonošském národním parku (GIS KRNAP Vrchlabí)
 Soubor map současného rozšíření lesních dřevin v Krkonošském národním parku (GIS KRNAP Vrchlabí) Mapa současného rozšíření borovice kleče v Krkonošském národním parku (GIS KRNAP Vrchlabí) Mapa současného
Soubor map současného rozšíření lesních dřevin v Krkonošském národním parku (GIS KRNAP Vrchlabí) Mapa současného rozšíření borovice kleče v Krkonošském národním parku (GIS KRNAP Vrchlabí) Mapa současného
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin
 Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace laboratorních úloh genetických předmětů metodikami pracujícími s ribonukleovými kyselinami pšenice Metodické návody pro laboratorní
Mendelova univerzita v Brně Agronomická fakulta Ústav biologie rostlin Inovace laboratorních úloh genetických předmětů metodikami pracujícími s ribonukleovými kyselinami pšenice Metodické návody pro laboratorní
Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae).
 Populační studie Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae). Biological Conservation 109: 249 258.
Populační studie Tomimatsu H. &OharaM. (2003): Genetic diversity and local population structure of fragmented populations of Trillium camschatcense (Trilliaceae). Biological Conservation 109: 249 258.
NOVELA ZÁKONA O OBCHODU S REPRODUKČNÍM MATERIÁLEM. Zákon č. 149/2003 Sb. ve znění zákona č. 232/2013 Sb.
 NOVELA ZÁKONA O OBCHODU S REPRODUKČNÍM MATERIÁLEM Zákon č. 149/2003 Sb. ve znění zákona č. 232/2013 Sb. MATERIÁLEM STÁVAJÍCÍ PRÁVNÍ ÚPRAVA Proč vůbec existuje něco jako zákon o obchodu s reprodukčním materiálem?
NOVELA ZÁKONA O OBCHODU S REPRODUKČNÍM MATERIÁLEM Zákon č. 149/2003 Sb. ve znění zákona č. 232/2013 Sb. MATERIÁLEM STÁVAJÍCÍ PRÁVNÍ ÚPRAVA Proč vůbec existuje něco jako zákon o obchodu s reprodukčním materiálem?
DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS
 DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS DETEKCE HOUBOVÝCH KONTAMINACÍ V PRÁŠKOVÉ PAPRICE POMOCÍ MOLEKULÁRNĚ BIOLOGICKÝCH METOD Trojan V., Hanáček P., Havel
DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS DETEKCE HOUBOVÝCH KONTAMINACÍ V PRÁŠKOVÉ PAPRICE POMOCÍ MOLEKULÁRNĚ BIOLOGICKÝCH METOD Trojan V., Hanáček P., Havel
Prokazování původu lesního reprodukčního materiálu pomocí genetických markerů
 Genetika a šlechtění lesních dřevin Prokazování původu lesního reprodukčního materiálu pomocí genetických markerů Ing. R. Longauer, CSc. Doc. Ing. RNDr. Eva Palátová, PhD. Ústav zakládání a pěstění lesů
Genetika a šlechtění lesních dřevin Prokazování původu lesního reprodukčního materiálu pomocí genetických markerů Ing. R. Longauer, CSc. Doc. Ing. RNDr. Eva Palátová, PhD. Ústav zakládání a pěstění lesů
Yi TPMT. Diagnostická souprava. Návod k použití. Haasova 27 Brno Česká republika. tel.:
 Yi TPMT Diagnostická souprava Návod k použití Výrobce: YBUX s.r.o. Haasova 27 Brno 616 00 Česká republika IČ 63487951 tel.: +420 541 423 710 e-mail: ybux@ybux.eu Název: Yi TPMT Popis: Diagnostická souprava
Yi TPMT Diagnostická souprava Návod k použití Výrobce: YBUX s.r.o. Haasova 27 Brno 616 00 Česká republika IČ 63487951 tel.: +420 541 423 710 e-mail: ybux@ybux.eu Název: Yi TPMT Popis: Diagnostická souprava
Mikrosatelity (STR, SSR, VNTR)
 Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
TESTOVÁNÍ GMO Praktikum fyziologie rostlin
 Teoretický úvod: TESTOVÁNÍ GMO Praktikum fyziologie rostlin 1 Teoretický úvod: TESTOVÁNÍ GMO Obecně na úvod Určitě jste už slyšeli pojem geneticky modifikovaný organismus (GMO). Úprava vlastností přirozeně
Teoretický úvod: TESTOVÁNÍ GMO Praktikum fyziologie rostlin 1 Teoretický úvod: TESTOVÁNÍ GMO Obecně na úvod Určitě jste už slyšeli pojem geneticky modifikovaný organismus (GMO). Úprava vlastností přirozeně
IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT GENELUTE)
 Strana 1 IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT GENELUTE) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku pomocí komerčního kitu GenElute. 2 Princip Proces izolace
Strana 1 IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT GENELUTE) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku pomocí komerčního kitu GenElute. 2 Princip Proces izolace
Využití molekulárních markerů v systematice a populační biologii rostlin. 12. Shrnutí,
 Využití molekulárních markerů v systematice a populační biologii rostlin 12. Shrnutí, Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length Polymorphism) založené
Využití molekulárních markerů v systematice a populační biologii rostlin 12. Shrnutí, Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length Polymorphism) založené
Seznam použitých chemikálií
 PŘÍLOHY Tabulky Tabulka č. 1. Přehled výsledků teplot tání pro amplifikáty primeru G3010A. Testování teplot tání rozředěného zásobího roztoku o koncentraci ndna 1ng / 1μl, ředění uvedeno v tabulce. Tabulka
PŘÍLOHY Tabulky Tabulka č. 1. Přehled výsledků teplot tání pro amplifikáty primeru G3010A. Testování teplot tání rozředěného zásobího roztoku o koncentraci ndna 1ng / 1μl, ředění uvedeno v tabulce. Tabulka
Mendelova genetika v příkladech. Genetické markery
 Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ
 INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
ÚJMA NA ŽIVOTNÍM PROSTŘEDÍ POŠKOZENÍM LESA
 Rožnovský, J., Litschmann, T. (ed.): XIV. Česko-slovenská bioklimatologická konference, Lednice na Moravě 2.-4. září 2002, ISBN 80-85813-99-8, s. 442-447 ÚJMA NA ŽIVOTNÍM PROSTŘEDÍ POŠKOZENÍM LESA Filip
Rožnovský, J., Litschmann, T. (ed.): XIV. Česko-slovenská bioklimatologická konference, Lednice na Moravě 2.-4. září 2002, ISBN 80-85813-99-8, s. 442-447 ÚJMA NA ŽIVOTNÍM PROSTŘEDÍ POŠKOZENÍM LESA Filip
8 PŘÍLOHA A - TABULKY
 8 PŘÍLOHA A - TABULKY Tabulka A - 1 Přehled reagencií a jejich koncentrací na přípravu reakční směsi PCR reagencie objem (µl) koncentrace ddh 2 O N x 7 PPP mix N x 10 5 primer N x 1 10 µm 3 primer N x
8 PŘÍLOHA A - TABULKY Tabulka A - 1 Přehled reagencií a jejich koncentrací na přípravu reakční směsi PCR reagencie objem (µl) koncentrace ddh 2 O N x 7 PPP mix N x 10 5 primer N x 1 10 µm 3 primer N x
CVD-T StripAssay. Kat. číslo 4-360. 20 testů 2-8 C
 CVD-T StripAssay Kat. číslo 4-360 20 testů 2-8 C Popis stripu Pracovní postup Izolace DNA Použijte čerstvou nebo zmraženou krev s EDTA nebo citrátem, jako antikoagulans, vyhněte se krvi s obsahem heparinu.
CVD-T StripAssay Kat. číslo 4-360 20 testů 2-8 C Popis stripu Pracovní postup Izolace DNA Použijte čerstvou nebo zmraženou krev s EDTA nebo citrátem, jako antikoagulans, vyhněte se krvi s obsahem heparinu.
Polymorfismus délky restrikčních fragmentů
 Polymorfismus délky restrikčních fragmentů Princip: Chemikálie: PCR produkt z předchozího praktického cvičení Endonukleáza KpnI 10 U μl -1 Pufr pro KpnI 10 koncentrovaný (Tris-HCl 100 mmol l -1 ph 7,5,
Polymorfismus délky restrikčních fragmentů Princip: Chemikálie: PCR produkt z předchozího praktického cvičení Endonukleáza KpnI 10 U μl -1 Pufr pro KpnI 10 koncentrovaný (Tris-HCl 100 mmol l -1 ph 7,5,
OBSAH 1 Úvod... 3 2 Uznané zdroje reprodukčního materiálu lesních dřevin... 5 3 Genové základny... 23
 OBSAH 1 Úvod... 3 2 Uznané zdroje reprodukčního materiálu lesních dřevin... 5 2.1 Rejstřík uznaných zdrojů reprodukčního materiálu... 5 2.1.1 Zdroje reprodukčního materiálu kategorie identifikovaný...
OBSAH 1 Úvod... 3 2 Uznané zdroje reprodukčního materiálu lesních dřevin... 5 2.1 Rejstřík uznaných zdrojů reprodukčního materiálu... 5 2.1.1 Zdroje reprodukčního materiálu kategorie identifikovaný...
TECHNICKÁ SPECIFIKACE Vybavení genetické laboratoře pro projekt EXTEMIT-K část B
 TECHNICKÁ SPECIFIKACE Vybavení genetické laboratoře pro projekt EXTEMIT-K část B OBSAH Sestava pro vertikální elektroforézu... 2 Jednotka pro elektroforézu... 3 Termocykler... 4 Elektrický zdroj pro elektroforézu...
TECHNICKÁ SPECIFIKACE Vybavení genetické laboratoře pro projekt EXTEMIT-K část B OBSAH Sestava pro vertikální elektroforézu... 2 Jednotka pro elektroforézu... 3 Termocykler... 4 Elektrický zdroj pro elektroforézu...
Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO.
 Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO. PCR Nucleotide Mix Předem připravený roztok ultračistých PCR deoxynukleotidů (datp,
Určeno pro obecné laboratorní užití. Není určeno pro použití v diagnostických postupech. POUZE PRO POUŽITÍ IN VITRO. PCR Nucleotide Mix Předem připravený roztok ultračistých PCR deoxynukleotidů (datp,
Soubor map struktury porostů na TVP v gradientu hory Plechý v Národním parku Šumava
 Soubor map struktury porostů na TVP v gradientu hory Plechý v Národním parku Šumava Vacek S., Remeš J., Bílek L., Vacek Z., Ulbrichová I. Soubor map: Mapa struktury porostu na TVP 12 v gradientu hory Plechý
Soubor map struktury porostů na TVP v gradientu hory Plechý v Národním parku Šumava Vacek S., Remeš J., Bílek L., Vacek Z., Ulbrichová I. Soubor map: Mapa struktury porostu na TVP 12 v gradientu hory Plechý
Fragment Analyzer UNIKÁTNÍ KAPILÁROVÝ FRAGMENTAČNÍ ANALYZÁTOR VÝBORNÉ VÝSLEDKY UNIKÁTNÍ VLASTNOSTI
 Fragment Analyzer UNIKÁTNÍ KAPILÁROVÝ FRAGMENTAČNÍ ANALYZÁTOR VÝBORNÉ VÝSLEDKY UNIKÁTNÍ VLASTNOSTI Výborný přístroj, výborné výsledky Fragmentační analyzátor je automatizovaný kapilárový systém s CE značkou
Fragment Analyzer UNIKÁTNÍ KAPILÁROVÝ FRAGMENTAČNÍ ANALYZÁTOR VÝBORNÉ VÝSLEDKY UNIKÁTNÍ VLASTNOSTI Výborný přístroj, výborné výsledky Fragmentační analyzátor je automatizovaný kapilárový systém s CE značkou
Neinvazivní genetika vlka obecného v Západních Karpatech Pavel Hulva, Barbora Černá Bolfíková, Milena Smetanová & Miroslav Kutal
 Neinvazivní genetika vlka obecného v Západních Karpatech Pavel Hulva, Barbora Černá Bolfíková, Milena Smetanová & Miroslav Kutal In: Kutal M. & Suchomel J., 2014: Analýza výskytu velkých šelem a průchodnosti
Neinvazivní genetika vlka obecného v Západních Karpatech Pavel Hulva, Barbora Černá Bolfíková, Milena Smetanová & Miroslav Kutal In: Kutal M. & Suchomel J., 2014: Analýza výskytu velkých šelem a průchodnosti
Mgr. et Mgr. Lenka Falková. Laboratoř agrogenomiky. Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita
 Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT NUCLEOSPIN FOOD)
 1252.1 Izolace DNA pro stanovení GMO metodou Strana 1 IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT NUCLEOSPIN FOOD) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku
1252.1 Izolace DNA pro stanovení GMO metodou Strana 1 IZOLACE DNA PRO STANOVENÍ GMO METODOU PCR (KIT NUCLEOSPIN FOOD) 1 Účel a rozsah Postup slouží k získání deoxyribonukleové kyseliny (DNA) ze vzorku
NÁVOD K POUŽITÍ PRO HER2 DNA QUANTIFICATION KIT
 IČ: 80 DIČ: CZ80 sales@intellmed.eu NÁVOD K POUŽITÍ PRO HER DNA QUANTIFICATION KIT IČ: 80 DIČ: CZ80 sales@intellmed.eu OBSAH Návod k použití pro HER DNA QUANTIFICATION KIT.... Úvod.... Označení.... Rozsah
IČ: 80 DIČ: CZ80 sales@intellmed.eu NÁVOD K POUŽITÍ PRO HER DNA QUANTIFICATION KIT IČ: 80 DIČ: CZ80 sales@intellmed.eu OBSAH Návod k použití pro HER DNA QUANTIFICATION KIT.... Úvod.... Označení.... Rozsah
Soubor map struktury porostů na TVP v oblasti Modravy v Národním parku Šumava
 Soubor map struktury porostů na TVP v oblasti Modravy v Národním parku Šumava Vacek S., Remeš J., Bílek L., Vacek Z., Ulbrichová I. Soubor map: Mapa struktury porostu na TVP 1 v oblasti Modravy v Národním
Soubor map struktury porostů na TVP v oblasti Modravy v Národním parku Šumava Vacek S., Remeš J., Bílek L., Vacek Z., Ulbrichová I. Soubor map: Mapa struktury porostu na TVP 1 v oblasti Modravy v Národním
LESY ČESKÉ REPUBLIKY, s. p. Ing. Zuzana Neznajová Semenářský závod v Týništi nad Orlicí
 LESY ČESKÉ REPUBLIKY, s. p. Ing. Zuzana Neznajová Semenářský závod v Týništi nad Orlicí Úloha SZ Týniště nad Orlicí ve schválené koncepci genetiky Vracíme české osivo zpět do českých lesů. 1 Semenářský
LESY ČESKÉ REPUBLIKY, s. p. Ing. Zuzana Neznajová Semenářský závod v Týništi nad Orlicí Úloha SZ Týniště nad Orlicí ve schválené koncepci genetiky Vracíme české osivo zpět do českých lesů. 1 Semenářský
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII. Martina Nováková, VŠCHT Praha
 MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna
 Mendelova genetika v příkladech Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna Roman Longauer, Ústav zakládání a pěstění lesů, MENDELU
Mendelova genetika v příkladech Populační genetika a fylogeneze jedle bělokoré analyzována pomocí izoenzymových genových markerů a variability mtdna Roman Longauer, Ústav zakládání a pěstění lesů, MENDELU
Metody inventarizace a hodnocení biodiverzity stromové složky
 ČESKÁ ZEMĚDĚLSKÁ UNIVERZITA V PRAZE Fakulta lesnická a dřevařská Metody inventarizace a hodnocení biodiverzity stromové složky Methods for inventory and biodiversity evaluation of tree layer SBORNÍK ZE
ČESKÁ ZEMĚDĚLSKÁ UNIVERZITA V PRAZE Fakulta lesnická a dřevařská Metody inventarizace a hodnocení biodiverzity stromové složky Methods for inventory and biodiversity evaluation of tree layer SBORNÍK ZE
QH71273. Výzkum nekrotrofních patogenů z r. Phytophthora na ekonomicky významných listnatých dřevinách
 QH71273 Výzkum nekrotrofních patogenů z r. Phytophthora na ekonomicky významných listnatých dřevinách Podprogram 1 Efektivní postupy v agrárním sektoru Výzkumný směr 12 - Ochrana zdraví rostlin a zvířat
QH71273 Výzkum nekrotrofních patogenů z r. Phytophthora na ekonomicky významných listnatých dřevinách Podprogram 1 Efektivní postupy v agrárním sektoru Výzkumný směr 12 - Ochrana zdraví rostlin a zvířat
Poznámky z přednášek HUL. Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.
 Poznámky z přednášek HUL Aktualizace zadávání a schvalování LHP LHP jako nástroj vlastníka. Historický vývoj LHP a jeho využití do současnosti. Význam LHP pro vlastníky lesů. Legislativní rámec LHP, zákony
Poznámky z přednášek HUL Aktualizace zadávání a schvalování LHP LHP jako nástroj vlastníka. Historický vývoj LHP a jeho využití do současnosti. Význam LHP pro vlastníky lesů. Legislativní rámec LHP, zákony
Jednotné pracovní postupy zkoušení krmiv STANOVENÍ OBSAHU MYKOTOXINŮ METODOU LC-MS - FUMONISIN B 1 A B 2
 Národní referenční laboratoř Strana 1 STANOVENÍ OBSAHU MYKOTOXINŮ METODOU LC-MS - FUMONISIN B 1 A B 2 1 Rozsah a účel Metoda je vhodná pro stanovení fumonisinů B 1 a B 2 v krmivech. 2 Princip Fumonisiny
Národní referenční laboratoř Strana 1 STANOVENÍ OBSAHU MYKOTOXINŮ METODOU LC-MS - FUMONISIN B 1 A B 2 1 Rozsah a účel Metoda je vhodná pro stanovení fumonisinů B 1 a B 2 v krmivech. 2 Princip Fumonisiny
Sure-MeDIP I. with magnetic beads and MNase. www.krd.cz
 Sure-MeDIP I with magnetic beads and MNase www.krd.cz 1 Obsah soupravy a skladování MeDIP souprava obsahuje reagencie na provedení 25 reakcí. Souprava je rozdělen do dvou částí, jedna je distribuována
Sure-MeDIP I with magnetic beads and MNase www.krd.cz 1 Obsah soupravy a skladování MeDIP souprava obsahuje reagencie na provedení 25 reakcí. Souprava je rozdělen do dvou částí, jedna je distribuována
Písemná zpráva zadavatele
 Písemná zpráva zadavatele veřejné zakázky zadávané dle zákona č. 137/2006 Sb., o veřejných zakázkách, ve znění účinném ke dni zahájení zadávacího řízení (dále jen ZVZ ). Veřejná zakázka Název: Ostatní
Písemná zpráva zadavatele veřejné zakázky zadávané dle zákona č. 137/2006 Sb., o veřejných zakázkách, ve znění účinném ke dni zahájení zadávacího řízení (dále jen ZVZ ). Veřejná zakázka Název: Ostatní
AFLP. protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech
 AFLP AFLP protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech AFLP protokoly kit Invitrogen bez použití kitu AmpliTaq Gold (ABI) možnost
AFLP AFLP protokoly standardizace AFLP hodnocení primárních dat dnes používané metody hodnocení fylogenetický signál v AFLP datech AFLP protokoly kit Invitrogen bez použití kitu AmpliTaq Gold (ABI) možnost
CYCLER CHECK. Test pro validaci teplotní uniformity cyklérů. Připraveno k použití, prealiquotováno. REF 7104 (10 testů) REF (4 testy)
 CZ Návod k použití CYCLER CHECK Test pro validaci teplotní uniformity cyklérů Připraveno k použití, prealiquotováno REF 7104 (10 testů) REF 71044 (4 testy) Obsah 1. Popis výrobku... 2 2. Materiál... 3
CZ Návod k použití CYCLER CHECK Test pro validaci teplotní uniformity cyklérů Připraveno k použití, prealiquotováno REF 7104 (10 testů) REF 71044 (4 testy) Obsah 1. Popis výrobku... 2 2. Materiál... 3
Laboratoř sekvenace DNA Servisní laboratoř biologické sekce PřF UK
 Laboratoř sekvenace DNA Založena 2003, momentálně jsme 2 zaměstnankyně, máme dvě spolupracovnice na DPP a jsme finančně soběstačné, včetně platů. Rok Počet sekvenací Počet fragmentací 2009 10 700 8 100
Laboratoř sekvenace DNA Založena 2003, momentálně jsme 2 zaměstnankyně, máme dvě spolupracovnice na DPP a jsme finančně soběstačné, včetně platů. Rok Počet sekvenací Počet fragmentací 2009 10 700 8 100
Využití molekulárních markerů v systematice a populační biologii rostlin. 2. Přehled aplikací a otázek
 Využití molekulárních markerů v systematice a populační biologii rostlin 2. Přehled aplikací a otázek Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length
Využití molekulárních markerů v systematice a populační biologii rostlin 2. Přehled aplikací a otázek Přehled molekulárních markerů 1. proteiny isozymy 2. DNA markery RFLP (Restriction Fragment Length
Kras XL StripAssay. Kat. číslo 5-680. 20 testů 2-8 C
 Kras XL StripAssay Kat. číslo 5-680 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Musí být použity vhodné metody extrakce DNA, v závislosti na typu vzorku, který má být vyšetřován. Doporučení
Kras XL StripAssay Kat. číslo 5-680 20 testů 2-8 C Popis stripu: Pracovní postup 1. Izolace DNA Musí být použity vhodné metody extrakce DNA, v závislosti na typu vzorku, který má být vyšetřován. Doporučení
POČET ROČNÍKŮ JEHLIC POPULACÍ BOROVICE LESNÍ. Needle year classes of Scots pine progenies. Jarmila Nárovcová. Abstract
 POČET ROČNÍKŮ JEHLIC POPULACÍ BOROVICE LESNÍ Needle year classes of Scots pine progenies Jarmila Nárovcová Výzkumný ústav lesního hospodářství a myslivosti, v. v. i. Výzkumná stanice Opočno Na Olivě 550
POČET ROČNÍKŮ JEHLIC POPULACÍ BOROVICE LESNÍ Needle year classes of Scots pine progenies Jarmila Nárovcová Výzkumný ústav lesního hospodářství a myslivosti, v. v. i. Výzkumná stanice Opočno Na Olivě 550
Potenciál a riziko využívání těžebních zbytků v borových porostech na majetku Městských lesů Doksy, s.r.o.
 Česká zemědělská univerzita v Praze Fakulta lesnická a dřevařská Soubor map Potenciál a riziko využívání těžebních zbytků v borových porostech na majetku Městských lesů Doksy, s.r.o. Doc. Ing. Jiří Remeš,
Česká zemědělská univerzita v Praze Fakulta lesnická a dřevařská Soubor map Potenciál a riziko využívání těžebních zbytků v borových porostech na majetku Městských lesů Doksy, s.r.o. Doc. Ing. Jiří Remeš,
Genetické markery, markery DNA
 Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
ANALYTICKÝ SYSTÉM PHOTOCHEM
 ANALYTICKÝ SYSTÉM PHOTOCHEM Analytický systém Photochem (firmy Analytik Jena, Německo) je vhodný pro stanovení celkové antioxidační kapacity (tj. celkové schopnosti vzorku vychytávat volné radikály) různých
ANALYTICKÝ SYSTÉM PHOTOCHEM Analytický systém Photochem (firmy Analytik Jena, Německo) je vhodný pro stanovení celkové antioxidační kapacity (tj. celkové schopnosti vzorku vychytávat volné radikály) různých
THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS
 THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS VÝBĚR NEJVHODNĚJŠÍHO ZPŮSOBU IZOLACE NUKLEOVÝCH KYSELIN NA ÚSTAVU MORFOLOGIE,
THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS VÝBĚR NEJVHODNĚJŠÍHO ZPŮSOBU IZOLACE NUKLEOVÝCH KYSELIN NA ÚSTAVU MORFOLOGIE,
2 Inkompatibilita v systému Rhesus. Upraveno z A.D.A.M.'s health encyclopedia
 2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
Určení koncentrace proteinu fluorescenční metodou v mikrotitračních destičkách
 Určení koncentrace proteinu fluorescenční metodou v mikrotitračních destičkách Teorie Stanovení celkových proteinů Celkové množství proteinů lze stanovit pomocí několika metod; například: Hartree-Lowryho
Určení koncentrace proteinu fluorescenční metodou v mikrotitračních destičkách Teorie Stanovení celkových proteinů Celkové množství proteinů lze stanovit pomocí několika metod; například: Hartree-Lowryho
Populačně-genetická data
 Populačně-genetická data Základy analýzy diploidních kodominantních znaků (Mendelovská dědičnost) mikrosatelity T jaderné sekvence (např. SSCP) CCGATCAATGCGGCAA G CCGATCACTGCGGCAA SNPs Typ získaných dat
Populačně-genetická data Základy analýzy diploidních kodominantních znaků (Mendelovská dědičnost) mikrosatelity T jaderné sekvence (např. SSCP) CCGATCAATGCGGCAA G CCGATCACTGCGGCAA SNPs Typ získaných dat
Seminář izolačních technologií
 Seminář izolačních technologií Zpracoval: Karel Bílek a Kateřina Svobodová Podpořeno FRVŠ 2385/2007 a 1305/2009 Úpravy a aktualizace: Pavla Chalupová ÚMFGZ MZLU v Brně 1 Lokalizace jaderné DNA 2 http://www.paternityexperts.com/basicgenetics.html
Seminář izolačních technologií Zpracoval: Karel Bílek a Kateřina Svobodová Podpořeno FRVŠ 2385/2007 a 1305/2009 Úpravy a aktualizace: Pavla Chalupová ÚMFGZ MZLU v Brně 1 Lokalizace jaderné DNA 2 http://www.paternityexperts.com/basicgenetics.html
Biologická Diversita. Různorodost druhů a genetická diversita uvnitř druhů
 Biologická Diversita Různorodost druhů a genetická diversita uvnitř druhů Sladkovodní ekosystémy 2.5% světových zásob vody je sladkovodních, z toho 99% led či podzemní voda Velká část využívaná pro zavlažování
Biologická Diversita Různorodost druhů a genetická diversita uvnitř druhů Sladkovodní ekosystémy 2.5% světových zásob vody je sladkovodních, z toho 99% led či podzemní voda Velká část využívaná pro zavlažování
Analýza DNA. Co zjišťujeme u DNA DNA. PCR polymerase chain reaction. Princip PCR PRINCIP METODY PCR
 o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED
 ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED FREKVENCE ALEL GENU KIT ASOCIOVANÉHO SE STRAKATOSTÍ TOBIANO U KONÍ PLEMENE PAINT HORSE Chalupová P., Déduchová
ALLELE FREQUENCY OF KIT GENE ASSOCIATED WITH TOBIANO SPOTTING PATTERN IN PAINT HORSE BREED FREKVENCE ALEL GENU KIT ASOCIOVANÉHO SE STRAKATOSTÍ TOBIANO U KONÍ PLEMENE PAINT HORSE Chalupová P., Déduchová
Péče o vnitrodruhovou diversitu na příkladu smrku v horských polohách. Antonín Jurásek, VS VÚLHM Opočno
 Péče o vnitrodruhovou diversitu na příkladu smrku v horských polohách Antonín Jurásek, VS VÚLHM Opočno 30.3.2016 Cíl Cílem je zjistit potenciální problémy a na základě dostupných poznatků výzkumu, stanovit
Péče o vnitrodruhovou diversitu na příkladu smrku v horských polohách Antonín Jurásek, VS VÚLHM Opočno 30.3.2016 Cíl Cílem je zjistit potenciální problémy a na základě dostupných poznatků výzkumu, stanovit
Stejskalová J., Kupka I.: Vliv lesních vegetačních stupňů na kvalitu semen jedle bělokoré... (ABIES ALBA MILL.) ABSTRACT
 VLIV LESNÍCH VEGETAČNÍCH STUPŇŮ NA KVALITU SEMEN JEDLE BĚLOKORÉ (ABIES ALBA MILL.) FOREST VEGETATION ZONES INFLUENCE ON SEED QUALITY OF SILVER FIR (ABIES ALBA MILL.).) JANA STEJSKALOVÁ, IVO KUPKA ABSTRACT
VLIV LESNÍCH VEGETAČNÍCH STUPŇŮ NA KVALITU SEMEN JEDLE BĚLOKORÉ (ABIES ALBA MILL.) FOREST VEGETATION ZONES INFLUENCE ON SEED QUALITY OF SILVER FIR (ABIES ALBA MILL.).) JANA STEJSKALOVÁ, IVO KUPKA ABSTRACT
