PLANT TRANSPOSABLE ELEMENTS AS A TOOL FOR LOCALIZATION
|
|
|
- Lucie Valentová
- před 7 lety
- Počet zobrazení:
Transkript
1 PLANT TRANSPOSABLE ELEMENTS AS A TOOL FOR LOCALIZATION GENE TRANSPOZONY ROSTLIN A JEJICH VYUŽITÍ PŘI LOKALIZACI GENŮ 1,2 Čegan R., 1,2 Hobza R., 2,3 Mráček J., 2 Kejnovský E., 4 Widmer A., 2 Vyskot B. 1 Department of Plant Biology, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemědělská 1, , Brno, Czech Republic 2 Plant Developmental Genetics, Institute of Biophysics, Academy of Sciences of the Czech Republic, Královopolská 135, , Brno, Czech Republic 3 Ústav lesnické botaniky, dendrologie a geobiocenologie, Lesnická a dřevařská fakulta, Mendelova zemědělská a lesnická univerzita v Brně, Zemědělská 3, BRNO, Česká republika 4 Plant Ecological Genetics, Institute of Integrative Biology, ETH Zurich, 8092 Zurich, Switzerland xcegan@node.mendelu.cz, qqhobza@node.mendelu.cz ABSTRACT Transposable elements (TE) and retroelements are frequently co-localized with heterochromatin and non-coding regions of genome. MITE elements (Miniature Inverted-repeats Transposable Elements) is a class of small TE without their own transposase. Surprisingly, MITEs are often found in the vicinity of genes in some model species (rice, maize). Mechanism of transposition of these elements to coding regions is still unclear. Plant genomes have often extremely huge size and are composed of mainly repetitive sequences. Many tools were designed to subtract coding DNA (genes) from rest of genomic DNA. In this paper, we focused on using MITE elements as a tool to find new genes in Silene latifolia genome. S. latifolia is a model plant for studies concerning sex chromosome evolution. Non-recombining chromosome Y is potentially affected by degeneration processes which reduce expression of Y linked genes. Previous methods used for identification of Y linked alleles enabled to identify only several candidates. We investigated distribution of some MITE elements in genome of S. latifolia in context of gene distribution. Based on GenBank data we identified a MITE element in S vulgaris. This species is closely related to S. latifolia and enabled us to characterize a homologue of the element in this species. We screened a BAC library of genomic clones with both MITE probe and complex probe containing previously published genes. We found strong co-localization of MITE element and genes in S. latifolia. We suggest to use MITE probe as a useful tool to find new genes in genomic libraries in S.latifolia and related species.
2 Key words: Transposable elements, MITEs, Silene species, BAC library, gene localization Acknowledgments: This project was supported by the Grant Agency of the Czech Republic (grant 204/05/H505) and Ministry of Education (grant LC06004).
3 ÚVOD Transpozony neboli mobilní genetické elementy tvoří pravidelnou součást genomů rostlin. Často navozují mutace genů, jsou odpovědny za přestavby chromozomů a můžou přenášet nové znaky (horizontální genový transfer). Transpozony obecně dělíme do dvou tříd. První třída jsou retroelementy a retrotranspozony a druhá třída jsou transponovatelné elementy. Retroelementy jsou transpozony pohybující se (množící se) v genomech systémem copy and paste tzn., že dochází k duplikaci pohybujícího se elementu. Hlavními zástupci této třídy I. jsou LTR (long terminal repeats) a non-ltr retroelementy. Transponovatelné elementy se dělí do tří skupin na autonomní elementy, neautonomní elementy a MITEs (Miniature Inverted-repeats Transposable Elements). Tyto elementy jsou v genomu přenášeny způsobem cut and paste tzn., že se z původního místa výskytu vystřihnou a včlení se na místo nové. MITE elementy jsou často hojně zastoupeny v genomech rostlin. Jejich původ je pravděpodobně odvozen od amplifikace neautonomních krátkých elementů, které ztratily schopnost samostatné transpozice, která je řízena transpozázou kódovanou příbuzným autonomním elementem (Zhang et al., 2001). V současnosti je známo několik rodin těchto elemetů: Stovaway (Bureau et Wessler, 1994a), Tourist (Bureau et Wessler, 1992), Spring (Han et Korban, 2007), mping (Nakazaki et al., 2003), Heartbreaker (Casa et al., 2000) a další. V genomu rýže představují největší podíl ze všech TE (Naito et al.,2006). Každý MITE element je tvořen koncovou invertovanou repeticí (TIRs Terminal Inverted Repeats), interní sekvencí a krátkou sekvencí, kterou je element ohraničen a kterou upřednostňuje ke své inzerci (TSD Target Site Duplication). Způsob jejich transpozice v genomu zatím nebyl objasněn, nicméně se předpokládá, že jejich transpozice je řízena transpozázou kódovanou příbuzným autonomním elementem, která specificky rozezná jejich koncové repetice (Casacuberta et Santiago, 2003). V současnosti jsou známé také aktivní MITE elemety, které se dosud v genomu transponují. Mnoho MITE elementů jsou nalézány v genově bohatých oblastech, nejčastěji v těsné blízkosti genů nebo přímo v intronech či exonech (Bureau et Wessler, 1994b; Bergero et al., 2008). MITE elementy ovlivňují transkripci a translaci genů, jejich transpozice může být také aktivována stresem a mají vliv na evoluci genů a genomů (Casacuberta et Santiago, 2003). Pokud jsou přítomny v pohlavních chromozomech můžou mít vliv na degeneraci chromozomu Y. Rostliny rodu Silene patří do čeledi Caryophyllaceae sekce Elisanthe. V rámci tohoto rodu můžeme mezi nimi najít mnoho zástupců s různým způsobem rozmnožování (asi 300 celkem). Většina druhů z tohoto rodu má 24 chromozomů. S. latifolia (Silenka širolistá) i S. dioica (Silenka dvoudomá) jsou dvoudomé (dioecické). Silene latifolia i S. dioica mají heteromorfní pohlavní chromozomy (XY), podobně jako člověk (Vyskot et Hobza, 2004). Rostliny Silene vulgaris jsou jednodomé (monoecické) nebo hermafroditní. Rod Silene je velice vhodný ke studiu: evoluce pohlavních chromozomů, rezistence k těžkým kovům,
4 cytoplazmatické samčí sterility a speciace. V dnešní době je známo mnoho metod, pomocí kterých se dají nalézt (identifikovat) nové geny a které byly v minulosti již použity (differential message display (Liang et Pardee, 1992), RNA fingerprinting by arbitrarily primed PCR (Welsh et al., 1992), cdna-aflp (Bachem et al., 1996), RDA - Representational Differential Analysis (Lisitsyn et al., 1993; Hubank et Schatz, 1994), SAGE - Serial Analysis of Gene Expression (Velculescu et al., 1995), mikrodisekce chromozomů (Hozier et al., 1996), IMAGE - integrated molecular analysis of genomes and their expression (Lennon, 1996), a další). My v této práci předkládáme další možný (alternativní) způsob jak nalézt nové geny za využití MITE elementů, o kterých je ze starších prací známo, že se nalézají v blízkosti genů či přímo v intronech nebo exonech. Tuto cestu jsme chtěli ověřit u rostlin z rodu Silene, speciálně u Silene latifolia. Na začátku této práce jsme si položili několik otázek: Nacházejí se MITE elementy také u rodu Silene? Pokud ano, v kterých částech genomu Silene se nacházejí? Kolokalizují MITE elementy s geny, podobně jako u jiných druhů? Jaká je hojnost těchto elementů v genomu? Vyskytují se tyto elementy v repetitivních či jedinečných sekvencích? Dají se MITE elementy využít ke hledání genů? A na tyto otázky se pokusíme odpovědět v následujícím příspěvku. MATERIÁL A METODIKA Rostlinný materiál a izolace DNA Rostliny Silene vulgaris, Silene dioica, Silene latifolia byly pěstovány ve skleníku za standardních podmínek. Genomická DNA byla izolována z mladých listů semenáčků pomocí DNAeasy Plant Mini Kitu (Qiagen). PCR protokol Standardní PCR podmínky byly 95 C po dobu 4 min, následovány 35 cykly 95 C/45s, 55 C/1 min, 72 C/5 min a finální inkubace 72 C/5 min. Pro amplifikaci MITE elementu byl odvozen primer 5 - TTATACTCCCTCCTATTCACCATTTT 3 ze sekvence MITE elementu z genu glutathione-s-transferázy Silene cucubalus, který byl nalezen prohledáváním již známých sekvencí genů Silene v databázích. Tento primer byl použit pro PCR s genomickou DNA S. vulgaris, S. latifolia a S. dioica. Klonování PCR produktů a sekvenování Po gelové elektroforéze (1% agaróza), byly PCR produkty přečištěny pomocí Qiagen PCR Purification Kitu a klonovány do pgem T-easy vectoru (Promega, Germeny). Purifikované vzorky byly sekvenovány v Institutu integrativní biologie v Curychu (ETH Zürich) pomocí 16 kapilárního sekvenátoru ABI 3100-Avant dle manuálu výrobce.
5 FISH (Fluorescence in situ hybridization) Mitotické chromozomy byly připraveny z kořenových špiček ošetřených podle Lengerové et al. (2004) s drobnými úpravami. Denaturace prób byla 10 minut při 75 C. Do hybridizační reakce bylo vzato ng denaturované próby a hybridizace probíhala 16 hodin při 37 C ve vlhké komůrce. DNA byla značena červeně pomocí Fluorolink Cy3-dUTP (Amersham Pharmacia Biotech) v kombinaci s nick translation mix (Roche) nebo zeleně se Spectrum Green direct-labeled dutp a Nick Translation kit (obojí Vysis). Chromozomy byly barveny DAPI (4, 6 - diamidino-2-phenylindole). Pozorování mitotických preparátů bylo provedeno fluorescenčním mikroskopem Olympus AX7O a snímky byly zpracovány pomocí programu ISIS. Screening BACové knihovny Silene latifolia Pro screening BACové knihovny S.latifolia byly použity PCR produkty 15 genů a PCR produkty našeho MITE elementu a elementů Bergero et al. (2008). PCR produkty byly vyříznuty z 1% agarózového gelu a přečištěny pomocí QIAquick Gel Extraction Kitu. Značení sond bylo provedeno pomocí Prime-It II Random Labeling Kitu a jako izotop byl použit α - 32 P datp, podle standardního protokolu. Membrány byly hybridizovány při 60 C po dobu 16 hodin a odmývány v 0,3x SSC/0.1 % SDS 20 minut a 0,1x SSC/0,1 % SDS 20 minut. Signály byly detekovány autoradiografií a vyhodnoceny podle manuálu dodavatele BACové knihovny. Southernova hybridizace Southernova hybridizace byla provedena dle Southerna (1975). Vybrané BACové klony, které pozitivně hybridizovaly s próbou obsahující mix 15 genů byly štěpeny restrikčním enzymem HindIII (NEB). Southernovou hybridizací byly přeneseny na membránu Hybond N+. Pro radioaktivní hybridizaci této membrány byl jako sonda použit PCR produkt MITE elementu. Radioaktivní hybridizace byla provedena stejně jak je uvedeno v odstavci Screening BACové knihovny Silene latifolia. VÝSLEDKY A DISKUZE Na počátku této práce u rodu Silene nebyl znám žádný MITE element. Proto jsme prohledali databáze sekvencí rodu Silene pomocí GenBank. V databázi jsme nalezli jeden MITE element poblíž 5 konce genu glutathione-s-transferázy u Silene cucubalus. Tento gen je pravděpodobně zodpovědný za rezistenci či toleranci k těžkým kovům (Prändl et Kutchan, 1992)(Silene cucubalus = Silene vulgaris). Pracovní název tohoto MITE elementu jsme zvolili MACAS (Mite As Coding Associated Sequence).
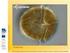
6 Sekvenování a složení MITE elementu Pomocí PCR a následného sekvenování jsme získali celkem 93 sekvencí MITE elementu ze S. latifolia, S.dioica, S. diclinis a S.vulgaris. Mezi jednotlivými druhy nebyl nalezen žádný rozdíl v sekvenci TIRs ani TSD (Tab. 1). Všechny sekvence se liší jen svou vnitřní částí. Název TIRs TSD délka bp Silene cucubalus glutathione- S-transferase gene MITE TACTCCCTCCCATCCTCTATTTTCTTCCCT TA 317 MITE element (MACAS) TCCCTCCTATTCACCATTTTCTT (23 bp) TA SlTO1 GGGGGTGTTTGGTT (14 bp) TTA 290 EITRI CTAGGTAGCAC (11 bp) CTCTTGAG 407 Tab.1 Přehled a porovnání základních vlastností MITE elementů v rodu Silene. (Pro srovnání jsou v tabulce zařazeny v současnosti již známé MITE elementy z rodu Silene (Bergero et al., 2008). Distribuce MITE elementu v genomu pomocí FISH Pomocí fluorescenční in situ hybridizace na metafázových chromozomech S.latifolia jsme zjistili, že náš MITE element je lokalizován v subtelomerových oblastech chromozomů (obr.1). Toto zjištění je v silné korelaci s výsledky získanými v naší laboratoři. Vyskot et al.(1999) zjistili, že genově bohaté oblasti jsou zvláště subtelomery chromozomů. Ve své práci využili tří přístupů. Byly to: studium acetylace histonů H4 metodou imunoprecipitace, studium DNA replikační kinetiky pomocí 5-bromdeoxyuridinu a cdna FISH.
7 Obr.1 FISH na metafázových chromozomech S. latifolia, červená barva (Cy3) MITE sonda, zelená barva (Spectrum green) sonda X.43.1(subtelomerová próba vhodná k rozlišení X a Y chromozomu), chromozomy barveny DAPI (4, 6 - diamidino-2-phenylindole) Screening BAC knihovny (Kolokalizují MITE elementy s geny, podobně jako u jiných druhů? Jaká je hojnost těchto elementů v genomu?) Pomocí prohledání (screening) BAC knihovny S.latifolia byly vybrány a izolovány BACy obsahující geny ze sondy (15) a jejich přítomnost ověřena pomocí PCR. Poté byly stejné membrány hybridizovány s MITE sondou. Bylo zjištěno, že MITE element je v genomu S. latifolia zastoupen přibližně v kopiích. Z 15 BACů obsahujících geny 11 obsahuje i MITE element. Tato skutečnost potvrzuje, že náš MITE element kolokalizuje s většinou námi použitých genů. Abychom pokryli celý genom S. latifolia jehož velikost je zhruba 3 x 10 9 bp potřebujeme alespoň BACů. Vezmeme-li v úvahu, že S. latifolia má přibližně genů a počet kopií našeho MITE elementu jsme stanovili na základě hybridizace na 1 000, tak to znamená, že jeden MITE element se nachází v každém 30 BACu. A uvědomíme-li si skutečnost, že více než polovina BACů obsahuje geny i MITE element, můžeme tvrdit, že geny jsou klastrovány. Vyskytují se tyto elementy v repetitivních či jedinečných oblastech genomu? Pro zodpovězení otázky, ve kterých oblastech genomu se náš MITE element nachází, zda to jsou repetitivní nebo jedinečné sekvence jsme provedli následující experiment. Vybrané BACy, u kterých bylo hybridizací zjištěno, že obsahují geny i MITE jsme štěpili restrikčním
8 enzymem HindIII a následně provedli Southernovu hybridizaci. Tato membrána byla nejprve hybridizována s genomickou DNA S. latifolia a poté s MITE elementem. Z obrázku 2 je zřejmé, že MITE elementy se nacházejí v nerepetitivních oblastech genomu a představují tak vhodný nástroj pro identifikaci unikátních markerů. Obr.2 Southernova hybridizace štěpených BACů a radioaktivní hybridizace s genomickou sondou a MITE elementem (L ladder 1kb, 1 štěpený BAC, 2 hybridizace s genomickou DNA S. latifolia, 3 hybridizace s MITE elementem) Další MITE elementy u rodu Silene V letošním roce byly publikovány a popsány první dva MITE elementy nalezené u rodu Silene (Bergero et al. 2008). V jejich práci jsou charakterizovány SlTO1 (Silene latifolia Touristlike element) a EITRI element. Pomocí našich přístupů jsme provedli některé experimenty zatím jen s SlTO1 elementem a zjistili jsme, že v genomu Silene latifolia má přibližně 400 kopií a nekolokalizuje s žádným genem, který byl použit ve výše uvedené studii přesto, že se nachází v intronu pohlavně vázaného genu SlCypX/Y (Bergero et al., 2007), který jsme v našich analýzách nepoužili, protože nebyl znám. Při FISH experimentech jsme získali pouze slabé signály na metafázových chromozomech Silene latifolia. Z těchto experimentů usuzujeme, že tento MITE element není tak hojný v genomu Silene latifolia, a není významně kolokalizován s genovými sekvencemi.
9 ZÁVĚR MITE elementy se v genomech rostlin často nacházejí v blízkosti genů. V této práci jsme identifikovali nový MITE element u druhu S. latifolia. Pomocí FISH jsem ukázali, že se tento element nachází v subtelomerách všech chromozomů. V dřívějších publikacích bylo ukázáno, že subtelomerické oblasti jsou u S. latifolia tvořeny převážně euchromatinem a jsou replikovány během buněčného cyklu jako první v pořadí. Výše uvedená data ukazují, že identifikovaný MITE element se v širším měřítku nachází poblíž genů. Přesnější určení vzdálenosti MITE elementů a genů bylo určeno pomocí paralelního prohledání BACové knihovny genomových fragmentů pomocí MITE sondy a směsné sondy obsahující geny. Bylo ukázáno, že MITE elementy jsou vzdáleny maximálně desítky kilobazí od kandidátních genů (kolokalizace MITE a genů v jednom BACu) a jsou inzerovány do unikátních (ne-repetivních) sekvencí (na základě Southernovy hybridizace štěpené BAC DNA s MITE a komplexní genomovou sondou). Výše uvedená data ukazují na vhodnost použití identifikovaného MITE elementu jako nástroje pro hledání nových markerů pro genetické mapování a hledání nových genů.
10 LITERATURA Bergero R., Forrest A., Kamau E. et Charlesworth D. (2007): Evolutionary strata on the X chromosomes of the dioecious plant Silene latifolia: evidence from new sex-linked genes. Genetics 175: Bergero R., Forrest A. et Charlesworth D. (2008): Active miniature transposons from a plant genome and its non-recombining Y chromosome. Genetics, 178: Bachem, C. W. B., Van der Hoeven S.R., de Bruijn S.R., Vreugdenhil D., Zabeau M. et Visser R.G.F. (1996): Visualisation of differential gene expression using a novel method of RNA fingerprinting based on AFLP: analysis of gene expression during tuber development. Plant J., 9: Bureau, T.E., Wessler, S.R. (1992): Tourist: A large family of small inverted repeat elements frequently associated with maize genes. Plant Cell, 4: Bureau, T.E., Wessler, S.R. (1994a): Stowaway: A new family of inverted repeat elements associated with the genes of both monocotyledonous and dicotyledonous plants. Plant Cell, 6: Bureau, T.E., Wessler, S.R. (1994b): Mobile inverted-repeat elements of the Tourist family are associated with the genes of many cereal grasses. PNAS, 91 (4): Casacuberta J. M., Santiago N. (2003): Plant LTR-retrotransposons and MITEs: control of transposition and impact on the evolutin of plant genes and genomes. Gene, 311: Casa M.A., Brouwer C., Nagel A., Wang L., Zhang Q., Kresowich S. et Wessler S.R. (2000): The MITE family Heartbreaker (Hbr): Molecular markers in maize. PNAS, 97 (18): Han Y., Korban S.S. (2007): Spring: A novel family of miniature inverted-repeat transposable elements is associated with genes in apple. Genomics, 90: Hozier J.C., Hall B.K., Sims K.R., Liechty M.C., Chen-Liu L. et Davis M.L. (1996): Chromosome Microdissection-Based Techniques for Genome Analysis. Methods, 9 (1): Hubank M., Schatz D.G. (1994): Identifying differences in mrna expression by representational difference analysis of cdna. Nucleic Acids Res. 22: Lengerová M., Kejnovský E., Hobza R., Macas J., Grant S.R. et.vyskot B. (2004): Multicolor FISH mapping of the dioecious model plant Silene latifolia. Theor Appl Genet, 108: Lennon G., Auffray C., Polymeropoulos M. et Soares M.B. (1996): The I.M.A.G.E. consorcium: an integrated molecular analysis of genomes and their expression. Genomics, 33: Liang P., Pardee A.B. (1992): Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction. Science, 257:
11 Lisitsyn N., Lisitsyn N. et Wigler M. (1993): Cloning the differences between two complex genomes. Science, 259: Naito K., Cho E., Yang G., Campbell M.A., Yano K., Okumuto Y., Tanisaka T. et Wessler S.R.. (2006): Dramatic amplification of a rice transposable element during recent domestication. PNAS, 103 (47): Nakazaki T., Okumuto Y., Horibata A., Yamahira S., Teraishi M., Nishida H., Inoue H. et Tanisaka T. (2003): Mobilization of a transposon in the rice genome. Nature, 421: Prändl R., Kutchan T. M. (1992): Nucleotide Sequence of the Gene for a Glutathione S- Transferase from Cell Suspension Cultures of Silene cucubalus. Plant Physiol., 99, Southern E.M. (1975): Detection of specific sequences among DNA fragments separated by gel electrophoresis. J Mol Biol., 98 (3): Velculescu V.E., Zhang L., Vogelstein B. et Kinzler K.W. (1995): Serial analysis of gene expression. Science, 270: Vyskot B., Siroky J., Hladilova R., Belyaev N.D. et Turner B.M. (1999): Euchromatic domains in plant chromosomes as revealed by H4 histone acetylation and early DNA replication. Genome, 42, (2): Vyskot B., Hobza R. (2004): Gender in plants: sex chromosomes are emerging from the fog. Trends in Genetics, 20: Welsh J., Chada K., Dalal S.S., Cheng R., Ralph D. et McClelland M. (1992): Arbitrarily primed PCR fingerprinting of RNA. Nucleic Acid Res., 20: Yang G., Zhang F., Hancock C.N., et Wessler S.R. (2007): Transposition of the rice miniature inverted repeat transposable element mping in Arabidopsis thaliana. PNAS, 104 (26): Zhang X., Jiang N., Feschotte C. et Wessler S.R. (2001): P instability factor: An active maize transposon system associated with the amplification of Tourist-like MITEs and a new superfamily of transposases. PNAS, 98 (22):
PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER
 PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER Trojan V., Hanáček P., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
PCR IN DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER Trojan V., Hanáček P., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University of Agriculture and Forestry in Brno, Zemedelska
Od sekvencí k chromozómům: výzkum repetitivní DNA rostlin v Laboratoři molekulární cytogenetiky BC AVČR
 ENBIK 2014 Od sekvencí k chromozómům: výzkum repetitivní DNA rostlin v Laboratoři molekulární cytogenetiky BC AVČR Jiří Macas Biologické centrum AVČR Ústav molekulární biologie rostlin České Budějovice
ENBIK 2014 Od sekvencí k chromozómům: výzkum repetitivní DNA rostlin v Laboratoři molekulární cytogenetiky BC AVČR Jiří Macas Biologické centrum AVČR Ústav molekulární biologie rostlin České Budějovice
Využití molekulárních markerů v systematice a populační biologii rostlin. 10. Další metody
 Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
Využití molekulárních markerů v systematice a populační biologii rostlin 10. Další metody Další molekulární markery trflp ISSRs (retro)transpozony kombinace a modifikace různých metod real-time PCR trflp
STUDY OF GENOME SIZE EVOLUTION
 STUDY OF GENOME SIZE EVOLUTION Čegan R. 1, 2, Obšívačová V. 1, 2, Kubeková H. 2, Kejnovský E. 2, Šafář J. 3, Vyskot B. 2, Hobza R. 1, 2 1 Department of Plant Biology, Faculty of Agronomy, Mendel University
STUDY OF GENOME SIZE EVOLUTION Čegan R. 1, 2, Obšívačová V. 1, 2, Kubeková H. 2, Kejnovský E. 2, Šafář J. 3, Vyskot B. 2, Hobza R. 1, 2 1 Department of Plant Biology, Faculty of Agronomy, Mendel University
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Poziční klonování Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s metodou pozičního klonování genů
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Poziční klonování Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s metodou pozičního klonování genů
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Metody používané v MB. analýza proteinů, nukleových kyselin
 Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Metody používané v MB. analýza proteinů, nukleových kyselin
 Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Metody používané v MB analýza proteinů, nukleových kyselin Nukleové kyseliny analýza a manipulace Elektroforéza (délka fragmentů, čistota, kvantifikace) Restrikční štěpení (manipulace s DNA, identifikace
Genové knihovny a analýza genomu
 Genové knihovny a analýza genomu Klonování genů Problém: genom organismů je komplexní a je proto obtížné v něm najít a klonovat specifický gen Klonování genů Po restrikčním štěpení genomové DNA pocházející
Genové knihovny a analýza genomu Klonování genů Problém: genom organismů je komplexní a je proto obtížné v něm najít a klonovat specifický gen Klonování genů Po restrikčním štěpení genomové DNA pocházející
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Využití DNA markerů ve studiu fylogeneze rostlin
 Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
Mendelova genetika v příkladech Využití DNA markerů ve studiu fylogeneze rostlin Ing. Petra VESELÁ Ústav lesnické botaniky, dendrologie a geobiocenologie LDF MENDELU Brno Tento projekt je spolufinancován
Struktura a analýza rostlinných genomů Jan Šafář
 Struktura a analýza rostlinných genomů Jan Šafář Ústav experimentální botaniky AV ČR, v.v.i Centrum regionu Haná pro biotechnologický a zemědělský výzkum Proč rostliny? Proč genom? Norman E. Borlaug Zelená
Struktura a analýza rostlinných genomů Jan Šafář Ústav experimentální botaniky AV ČR, v.v.i Centrum regionu Haná pro biotechnologický a zemědělský výzkum Proč rostliny? Proč genom? Norman E. Borlaug Zelená
Inovace studia molekulární a buněčné biologie
 Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. MBIO1/Molekulární biologie 1 Tento projekt je spolufinancován
Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. MBIO1/Molekulární biologie 1 Tento projekt je spolufinancován
Metody používané v MB. analýza proteinů, nukleových kyselin
 Metody používané v MB analýza proteinů, nukleových kyselin Proteiny analýza a manipulace Izolace, purifikace (rozdělovací metody) Centrifugace Chromatografie Elektroforéza Blotting (identifikace, western
Metody používané v MB analýza proteinů, nukleových kyselin Proteiny analýza a manipulace Izolace, purifikace (rozdělovací metody) Centrifugace Chromatografie Elektroforéza Blotting (identifikace, western
THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS
 THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS VÝBĚR NEJVHODNĚJŠÍHO ZPŮSOBU IZOLACE NUKLEOVÝCH KYSELIN NA ÚSTAVU MORFOLOGIE,
THE CHOICE OF THE MOST SUITABLE TECHNIQUE FOR ISOLATION OF NUCLEIC ACIDS AT DEPARTMENT OF ANIMAL MORPHOLOGY, PHYSIOLOGY AND GENETICS VÝBĚR NEJVHODNĚJŠÍHO ZPŮSOBU IZOLACE NUKLEOVÝCH KYSELIN NA ÚSTAVU MORFOLOGIE,
ISOLATION OF PHOSPHOPROTEOM AND ITS APPLICATION IN STUDY OF THE EFFECT OF CYTOKININ ON PLANTS
 ISOLATION OF PHOSPHOPROTEOM AND ITS APPLICATION IN STUDY OF THE EFFECT OF CYTOKININ ON PLANTS IZOLACE FOSFOPROTEOMU A JEHO VYUŽITÍ PŘI STUDIU ÚČINKU CYTOKININŮ NA ROSTLINU Černý M., Brzobohatý B. Department
ISOLATION OF PHOSPHOPROTEOM AND ITS APPLICATION IN STUDY OF THE EFFECT OF CYTOKININ ON PLANTS IZOLACE FOSFOPROTEOMU A JEHO VYUŽITÍ PŘI STUDIU ÚČINKU CYTOKININŮ NA ROSTLINU Černý M., Brzobohatý B. Department
ISSR (Inter simple sequence repeat) Jiří Košnar
 ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
ISSR (Inter simple sequence repeat) Jiří Košnar Přírodovědecká fakulta Jihočeské univerzity v Č. Budějovicích HISTORIE relativně nová metoda: Zietkiewicz E., Rafalski A., Labuda D. (1994): Genome fingerprinting
Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci. reg. č.: CZ.1.07/2.2.00/
 Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci reg. č.: CZ.1.07/2.2.00/28.0088 Hybridizační metody v diagnostice Mgr. Gabriela Kořínková, Ph.D. Laboratoř molekulární
Implementace laboratorní medicíny do systému vzdělávání na Univerzitě Palackého v Olomouci reg. č.: CZ.1.07/2.2.00/28.0088 Hybridizační metody v diagnostice Mgr. Gabriela Kořínková, Ph.D. Laboratoř molekulární
Využití průtokové cytometrie v analýze savčích chromozomů
 Využití průtokové cytometrie v analýze savčích chromozomů Jan Fröhlich frohlich@vri.cz Brno 17.-18.5.2015 Flowcytometrické karyotypování a sorting chromozomů - Historie Užívána od pol. 70. let 20.stol
Využití průtokové cytometrie v analýze savčích chromozomů Jan Fröhlich frohlich@vri.cz Brno 17.-18.5.2015 Flowcytometrické karyotypování a sorting chromozomů - Historie Užívána od pol. 70. let 20.stol
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION
 USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
USING OF AUTOMATED DNA SEQUENCING FOR PORCINE CANDIDATE GENES POLYMORFISMS DETECTION VYUŽITÍ AUTOMATICKÉHO SEKVENOVÁNÍ DNA PRO DETEKCI POLYMORFISMŮ KANDIDÁTNÍCH GENŮ U PRASAT Vykoukalová Z., Knoll A.,
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Analýza transkriptomu Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s moderními metodami komplexní
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Analýza transkriptomu Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s moderními metodami komplexní
DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS
 DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS DETEKCE HOUBOVÝCH KONTAMINACÍ V PRÁŠKOVÉ PAPRICE POMOCÍ MOLEKULÁRNĚ BIOLOGICKÝCH METOD Trojan V., Hanáček P., Havel
DETECTION OF FUNGAL CONTAMINATIONS IN POWDERED PEPPER USING MOLECULAR BIOLOGICAL METHODS DETEKCE HOUBOVÝCH KONTAMINACÍ V PRÁŠKOVÉ PAPRICE POMOCÍ MOLEKULÁRNĚ BIOLOGICKÝCH METOD Trojan V., Hanáček P., Havel
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Fyzické mapování Fyzické kontigové mapy Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s konstrukcí
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Fyzické mapování Fyzické kontigové mapy Ing. Hana Šimková, CSc. Cíl přednášky - seznámení s konstrukcí
Determinanty lokalizace nukleosomů
 METODY STUDIA CHROMATINU Topologie DNA a nukleosomů Struktura nukleosomu 1.65-1.8 otáčky Struktura nukleosomu 10.5 nt 1.8 otáčky 10n, 10n + 5 146 nt Determinanty lokalizace nukleosomů mechanické vlastnosti
METODY STUDIA CHROMATINU Topologie DNA a nukleosomů Struktura nukleosomu 1.65-1.8 otáčky Struktura nukleosomu 10.5 nt 1.8 otáčky 10n, 10n + 5 146 nt Determinanty lokalizace nukleosomů mechanické vlastnosti
Genetické mapování jako nástroj ke studiu evoluce pohlavních chromosomů rodu Silene
 BIOLOGICKÉ LISTY 68 (3): 183-189, 2003 Genetické mapování jako nástroj ke studiu evoluce pohlavních chromosomů rodu Silene VLADIMÍRA HYKELOVÁ Biofyzikální ústav AV ČR, Laboratoř vývojové genetiky rostlin,
BIOLOGICKÉ LISTY 68 (3): 183-189, 2003 Genetické mapování jako nástroj ke studiu evoluce pohlavních chromosomů rodu Silene VLADIMÍRA HYKELOVÁ Biofyzikální ústav AV ČR, Laboratoř vývojové genetiky rostlin,
Sekvenování nové generace. Radka Reifová
 Sekvenování nové generace Radka Reifová Prezentace ke stažení www.natur.cuni.cz/zoologie/biodiversity v záložce Přednášky 1. Přehled sekvenačních metod nové generace 2. Využití sekvenačních metod nové
Sekvenování nové generace Radka Reifová Prezentace ke stažení www.natur.cuni.cz/zoologie/biodiversity v záložce Přednášky 1. Přehled sekvenačních metod nové generace 2. Využití sekvenačních metod nové
Výuka genetiky na Přírodovědecké fakultě UK v Praze
 Výuka genetiky na Přírodovědecké fakultě UK v Praze Studium biologie na PřF UK v Praze Bakalářské studijní programy / obory Biologie Biologie ( duhový bakalář ) Ekologická a evoluční biologie ( zelený
Výuka genetiky na Přírodovědecké fakultě UK v Praze Studium biologie na PřF UK v Praze Bakalářské studijní programy / obory Biologie Biologie ( duhový bakalář ) Ekologická a evoluční biologie ( zelený
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Hybridizace nukleových kyselin
 Hybridizace nukleových kyselin Tvorba dvouřetězcových hybridů za dvou jednořetězcových a komplementárních molekul Založena na schopnosti denaturace a renaturace DNA. Denaturace DNA oddělení komplementárních
Hybridizace nukleových kyselin Tvorba dvouřetězcových hybridů za dvou jednořetězcových a komplementárních molekul Založena na schopnosti denaturace a renaturace DNA. Denaturace DNA oddělení komplementárních
Transpozony - mobilní genetické elementy
 Transpozony - mobilní genetické elementy Tvoří pravidelnou součást genomu prokaryot i eukaryot (až 50% genomu) Navozují mutace genů (inzerční inaktivace, polární mutace, změny exprese genů) Jsou zodpovědné
Transpozony - mobilní genetické elementy Tvoří pravidelnou součást genomu prokaryot i eukaryot (až 50% genomu) Navozují mutace genů (inzerční inaktivace, polární mutace, změny exprese genů) Jsou zodpovědné
Genotypování: Využití ve šlechtění a určení identity odrůd
 Molekulární přístupy ve šlechtění rostlin Aplikovaná genomika Genotypování: Využití ve šlechtění a určení identity odrůd Miroslav Valárik 14.2. 2017 Šlěchtění rostlin: Cílený výběr a manipulace s genomy
Molekulární přístupy ve šlechtění rostlin Aplikovaná genomika Genotypování: Využití ve šlechtění a určení identity odrůd Miroslav Valárik 14.2. 2017 Šlěchtění rostlin: Cílený výběr a manipulace s genomy
Výzkumné centrum genomiky a proteomiky. Ústav experimentální medicíny AV ČR, v.v.i.
 Výzkumné centrum genomiky a proteomiky Ústav experimentální medicíny AV ČR, v.v.i. Systém pro sekvenování Systém pro čipovou analýzu Systém pro proteinovou analýzu Automatický sběrač buněk Systém pro sekvenování
Výzkumné centrum genomiky a proteomiky Ústav experimentální medicíny AV ČR, v.v.i. Systém pro sekvenování Systém pro čipovou analýzu Systém pro proteinovou analýzu Automatický sběrač buněk Systém pro sekvenování
Hybridizace. doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz
 Hybridizace doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2013 Obsah přednášky 1) Způsoby provedení hybridizace 2) Hybridizace v roztoku 3) Příprava značených sond 4) Hybridizace
Hybridizace doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2013 Obsah přednášky 1) Způsoby provedení hybridizace 2) Hybridizace v roztoku 3) Příprava značených sond 4) Hybridizace
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII. Martina Nováková, VŠCHT Praha
 MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
MOLEKULÁRNĚ BIOLOGICKÉ METODY V ENVIRONMENTÁLNÍ MIKROBIOLOGII Martina Nováková, VŠCHT Praha MOLEKULÁRNÍ BIOLOGIE V BIOREMEDIACÍCH enumerace FISH průtoková cytometrie klonování produktů PCR sekvenování
Metody detekce poškození DNA
 STABILITA GENOMU II. Metody detekce poškození DNA Metody detekce poškození DNA Možnosti stanovení: 1. poškození DNA per se nebo 2. jeho následky mutace genů a mutace chromosomů 1. Detekce poškození DNA
STABILITA GENOMU II. Metody detekce poškození DNA Metody detekce poškození DNA Možnosti stanovení: 1. poškození DNA per se nebo 2. jeho následky mutace genů a mutace chromosomů 1. Detekce poškození DNA
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Metody molekulární biologie
 Metody molekulární biologie 1. Základní metody molekulární biologie A. Izolace nukleových kyselin Metody využívající různé rozpustnosti Metody adsorpční Izolace RNA B. Centrifugační techniky o Princip
Metody molekulární biologie 1. Základní metody molekulární biologie A. Izolace nukleových kyselin Metody využívající různé rozpustnosti Metody adsorpční Izolace RNA B. Centrifugační techniky o Princip
Charakterizace hybridních trav pomocí cytogenetických a molekulárních metod
 Molekulární přístupy ve šlechtění rostlin Olomouc 14. února, 2017 Charakterizace hybridních trav pomocí cytogenetických a molekulárních metod Jan Bartoš Ústav experimentální botaniky Olomouc, Czech Republic
Molekulární přístupy ve šlechtění rostlin Olomouc 14. února, 2017 Charakterizace hybridních trav pomocí cytogenetických a molekulárních metod Jan Bartoš Ústav experimentální botaniky Olomouc, Czech Republic
UNIVERZITA PARDUBICE FAKULTA CHEMICKO-TECHNOLOGICKÁ DISERTAČNÍ PRÁCE
 UNIVERZITA PARDUBICE FAKULTA CHEMICKO-TECHNOLOGICKÁ DISERTAČNÍ PRÁCE 2009 Mgr. Petra Horáková UNIVERZITA PARDUBICE FAKULTA CHEMICKO-TECHNOLOGICKÁ Katedra analytické chemie Elektrochemická analýza nukleotidových
UNIVERZITA PARDUBICE FAKULTA CHEMICKO-TECHNOLOGICKÁ DISERTAČNÍ PRÁCE 2009 Mgr. Petra Horáková UNIVERZITA PARDUBICE FAKULTA CHEMICKO-TECHNOLOGICKÁ Katedra analytické chemie Elektrochemická analýza nukleotidových
Genetické markery, markery DNA
 Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
Obecná genetika Genetické markery, markery DNA Prof. Ing. Dušan GÖMÖRY, DrSc. Ing. Roman LONGAUER, CSc. Ústav zakládání a pěstění lesů LDF MENDELU Brno Tento projekt je spolufinancován Evropským sociálním
MOBILNÍ GENETICKÉ ELEMENTY. Lekce 13 kurzu GENETIKA Doc. RNDr. Jindřich Bříza, CSc.
 MOBILNÍ GENETICKÉ ELEMENTY Lekce 13 kurzu GENETIKA Doc. RNDr. Jindřich Bříza, CSc. Demerec (1937) popsal nestabilní mutace u D. melanogaster B. McClintocková (1902-1992, Nobelova cena 1983) ukázala ve
MOBILNÍ GENETICKÉ ELEMENTY Lekce 13 kurzu GENETIKA Doc. RNDr. Jindřich Bříza, CSc. Demerec (1937) popsal nestabilní mutace u D. melanogaster B. McClintocková (1902-1992, Nobelova cena 1983) ukázala ve
Klonování DNA a fyzikální mapování genomu
 Klonování DNA a fyzikální mapování genomu. Terminologie Klonování je proces tvorby klonů Klon je soubor identických buněk (příp. organismů) odvozených ze společného předka dělením (např. jedna bakteriální
Klonování DNA a fyzikální mapování genomu. Terminologie Klonování je proces tvorby klonů Klon je soubor identických buněk (příp. organismů) odvozených ze společného předka dělením (např. jedna bakteriální
Bakteriální transpozony
 Bakteriální transpozony Transpozon = sekvence DNA schopná transpozice, tj. přemístění z jednoho místa v genomu do jiného místa Transpozice = proces přemístění transpozonu Transponáza (transpozáza) = enzym
Bakteriální transpozony Transpozon = sekvence DNA schopná transpozice, tj. přemístění z jednoho místa v genomu do jiného místa Transpozice = proces přemístění transpozonu Transponáza (transpozáza) = enzym
Metody studia exprese mrna. jádro a genová exprese 2007
 Metody studia exprese mrna Buněčné jádro a genová exprese 2007 Aktivita genu je primárn ě vyjád ř ena jeho transkripcí-prvním krokem vedoucím k syntéze kódovaného proteinu. Cíle metod Ur č ení mno ž ství
Metody studia exprese mrna Buněčné jádro a genová exprese 2007 Aktivita genu je primárn ě vyjád ř ena jeho transkripcí-prvním krokem vedoucím k syntéze kódovaného proteinu. Cíle metod Ur č ení mno ž ství
Struktura a organizace genomů
 CG020 Genomika Přednáška 8 Struktura a organizace genomů Markéta Pernisová Funkční genomika a proteomika rostlin, Mendelovo centrum genomiky a proteomiky rostlin, Středoevropský technologický institut
CG020 Genomika Přednáška 8 Struktura a organizace genomů Markéta Pernisová Funkční genomika a proteomika rostlin, Mendelovo centrum genomiky a proteomiky rostlin, Středoevropský technologický institut
 KLASICKÉ A MOLEKULÁRN RNÍ METODY V KLINICKÉ A ONKOLOGICKÉ CYTOGENETICE. Zuzana Zemanová CENTRUM NÁDOROVN DOROVÉ CYTOGENETIKY Ústav klinické biochemie a laboratorní diagnostiky VFN a 1. LF UK PRAHA CYTOGENETIKA
KLASICKÉ A MOLEKULÁRN RNÍ METODY V KLINICKÉ A ONKOLOGICKÉ CYTOGENETICE. Zuzana Zemanová CENTRUM NÁDOROVN DOROVÉ CYTOGENETIKY Ústav klinické biochemie a laboratorní diagnostiky VFN a 1. LF UK PRAHA CYTOGENETIKA
Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis.
 Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
Populační studie Kameyama Y. et al. (2001): Patterns and levels of gene flow in Rhododendron metternichii var. hondoense revealed by microsatellite analysis. Molecular Ecology 10:205 216 Proč to studovali?
Mgr. et Mgr. Lenka Falková. Laboratoř agrogenomiky. Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita
 Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
Mgr. et Mgr. Lenka Falková Laboratoř agrogenomiky Ústav morfologie, fyziologie a genetiky zvířat Mendelova univerzita 9. 9. 2015 Šlechtění Užitek hospodářská zvířata X zájmová zvířata Zemědělství X chovatelství
O původu života na Zemi Václav Pačes
 O původu života na Zemi Václav Pačes Ústav molekulární genetiky Akademie věd ČR centrální dogma replikace transkripce DNA RNA protein reverzní transkripce translace informace funkce Exon 1 Intron (413
O původu života na Zemi Václav Pačes Ústav molekulární genetiky Akademie věd ČR centrální dogma replikace transkripce DNA RNA protein reverzní transkripce translace informace funkce Exon 1 Intron (413
Návrh směrnic pro správnou laboratorní diagnostiku Friedreichovy ataxie.
 Návrh směrnic pro správnou laboratorní diagnostiku Friedreichovy ataxie. Připravila L.Fajkusová Online Mendelian Inheritance in Man: #229300 FRIEDREICH ATAXIA 1; FRDA *606829 FRDA GENE; FRDA Popis onemocnění
Návrh směrnic pro správnou laboratorní diagnostiku Friedreichovy ataxie. Připravila L.Fajkusová Online Mendelian Inheritance in Man: #229300 FRIEDREICH ATAXIA 1; FRDA *606829 FRDA GENE; FRDA Popis onemocnění
MOLEKULÁRNÍ BIOLOGIE. 2. Polymerázová řetězová reakce (PCR)
 MOLEKULÁRNÍ BIOLOGIE 2. Polymerázová řetězová reakce (PCR) Náplň praktik 1. Izolace DNA z buněk bukální sliznice - izolační kit MACHEREY-NAGEL 2. PCR polymerázová řetězová reakce (templát gdna) 3. Restrikční
MOLEKULÁRNÍ BIOLOGIE 2. Polymerázová řetězová reakce (PCR) Náplň praktik 1. Izolace DNA z buněk bukální sliznice - izolační kit MACHEREY-NAGEL 2. PCR polymerázová řetězová reakce (templát gdna) 3. Restrikční
EFFECT OF CADMIUM ON TOBACCO CELL SUSPENSION BY-2
 EFFECT OF CADMIUM ON TOBACCO CELL SUSPENSION BY-2 Štěpán Z., Klemš M., Zítka O., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University in Brno, Zemědělská 1, 613 00 Brno, Czech Republic
EFFECT OF CADMIUM ON TOBACCO CELL SUSPENSION BY-2 Štěpán Z., Klemš M., Zítka O., Havel L. Department of Plant Biology, Faculty of Agronomy, Mendel University in Brno, Zemědělská 1, 613 00 Brno, Czech Republic
NGS analýza dat. kroužek, Alena Musilová
 NGS analýza dat kroužek, 16.12.2016 Alena Musilová Typy NGS experimentů Název Materiál Cílí na..? Cíl experimentu? amplikon DNA malý počet vybraných genů hledání variant exom DNA všechny geny hledání
NGS analýza dat kroužek, 16.12.2016 Alena Musilová Typy NGS experimentů Název Materiál Cílí na..? Cíl experimentu? amplikon DNA malý počet vybraných genů hledání variant exom DNA všechny geny hledání
Crossing-over. over. synaptonemální komplex
 Genetické mapy Crossing-over over v průběhu profáze I meiózy princip rekombinace genetického materiálu mezi maternálním a paternálním chromosomem synaptonemální komplex zlomy a nová spojení chromatinových
Genetické mapy Crossing-over over v průběhu profáze I meiózy princip rekombinace genetického materiálu mezi maternálním a paternálním chromosomem synaptonemální komplex zlomy a nová spojení chromatinových
Mgr. Veronika Peňásová vpenasova@fnbrno.cz Laboratoř molekulární diagnostiky, OLG FN Brno Klinika dětské onkologie, FN Brno
 Retinoblastom Mgr. Veronika Peňásová vpenasova@fnbrno.cz Laboratoř molekulární diagnostiky, OLG FN Brno Klinika dětské onkologie, FN Brno Retinoblastom (RBL) zhoubný nádor oka, pocházející z primitivních
Retinoblastom Mgr. Veronika Peňásová vpenasova@fnbrno.cz Laboratoř molekulární diagnostiky, OLG FN Brno Klinika dětské onkologie, FN Brno Retinoblastom (RBL) zhoubný nádor oka, pocházející z primitivních
6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života?
 6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života? Pamatujete na to, co se objevilo v pracích Charlese Darwina a Alfreda Wallace ohledně vývoje druhů? Aby mohl mechanismus přírodního
6. Kde v DNA nalézáme rozdíly, zodpovědné za obrovskou diverzitu života? Pamatujete na to, co se objevilo v pracích Charlese Darwina a Alfreda Wallace ohledně vývoje druhů? Aby mohl mechanismus přírodního
Některé vlastnosti DNA důležité pro analýzu
 Některé vlastnosti DNA důležité pro analýzu Spiralizace Denaturace Záporný náboj Syntéza Ligace Rekombinace Mutabilita Despiralizace Reasociace Štěpení Metody používané k analýze DNA Southern blotting
Některé vlastnosti DNA důležité pro analýzu Spiralizace Denaturace Záporný náboj Syntéza Ligace Rekombinace Mutabilita Despiralizace Reasociace Štěpení Metody používané k analýze DNA Southern blotting
GENETIKA A MOLEKULÁRNĚ GENETICKÁ DIAGNOSTIKA DUCHENNEOVY MUSKULÁRNÍ DYSTROFIE
 GENETIKA A MOLEKULÁRNĚ GENETICKÁ DIAGNOSTIKA DUCHENNEOVY MUSKULÁRNÍ DYSTROFIE POHLED Z LABORATOŘE Petra Hedvičákov ková ÚBLG FN Motol Odd. lékal kařské molekulárn rní genetiky MZO 00064203 23.-24.5.2008
GENETIKA A MOLEKULÁRNĚ GENETICKÁ DIAGNOSTIKA DUCHENNEOVY MUSKULÁRNÍ DYSTROFIE POHLED Z LABORATOŘE Petra Hedvičákov ková ÚBLG FN Motol Odd. lékal kařské molekulárn rní genetiky MZO 00064203 23.-24.5.2008
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Mendelova genetika v příkladech. Genetické markery
 Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
Mendelova genetika v příkladech Genetické markery Tento projekt je spolufinancován Evropským sociálním fondem a Státním rozpočtem ČR InoBio CZ.1.07/2.2.00/28.0018 Hodnocení genetické proměnlivosti Fenotypový
Referenční lidský genom. Rozdíly v genomové DNA v lidské populaci. Odchylky od referenčního genomu. Referenční lidský genom.
 Referenční lidský genom Rozdíly v genomové DNA v lidské populaci Zdroj DNA: 60% sekvencí pochází ze sekvenování DNA od jednoho dárce (sekvenování a sestavování BAC klonů) některá místa genomu se nepodařilo
Referenční lidský genom Rozdíly v genomové DNA v lidské populaci Zdroj DNA: 60% sekvencí pochází ze sekvenování DNA od jednoho dárce (sekvenování a sestavování BAC klonů) některá místa genomu se nepodařilo
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/
 Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Sekvenování genomů Ing. Hana Šimková, CSc. Cíl přednášky - seznámení se strategiemi celogenomového sekvenování,
Inovace studia molekulární a buněčné biologie reg. č. CZ.1.07/2.2.00/07.0354 Genomika (KBB/GENOM) Sekvenování genomů Ing. Hana Šimková, CSc. Cíl přednášky - seznámení se strategiemi celogenomového sekvenování,
Molekulární genetika II zimní semestr 4. výukový týden ( )
 Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika II zimní semestr 4. výukový týden (27.10. 31.10.2008) prenatální DNA diagnostika presymptomatická Potvrzení diagnózy Diagnostika
Ústav biologie a lékařské genetiky 1.LF UK a VFN, Praha Molekulární genetika II zimní semestr 4. výukový týden (27.10. 31.10.2008) prenatální DNA diagnostika presymptomatická Potvrzení diagnózy Diagnostika
Typy nukleových kyselin. deoxyribonukleová (DNA); ribonukleová (RNA).
 Typy nukleových kyselin Existují dva typy nukleových kyselin (NA, z anglických slov nucleic acid): deoxyribonukleová (DNA); ribonukleová (RNA). DNA je lokalizována v buněčném jádře, RNA v cytoplasmě a
Typy nukleových kyselin Existují dva typy nukleových kyselin (NA, z anglických slov nucleic acid): deoxyribonukleová (DNA); ribonukleová (RNA). DNA je lokalizována v buněčném jádře, RNA v cytoplasmě a
Sekvenování nové generace. Radka Reifová
 Sekvenování nové generace Radka Reifová Prezentace ke stažení www.natur.cuni.cz/zoologie/biodiversity v záložce Přednášky 1. Přehled sekvenačních metod nové generace 2. Využití sekvenačních metod nové
Sekvenování nové generace Radka Reifová Prezentace ke stažení www.natur.cuni.cz/zoologie/biodiversity v záložce Přednášky 1. Přehled sekvenačních metod nové generace 2. Využití sekvenačních metod nové
Centrum aplikované genomiky, Ústav dědičných metabolických poruch, 1.LFUK
 ové technologie v analýze D A, R A a proteinů Stanislav Kmoch Centrum aplikované genomiky, Ústav dědičných metabolických poruch, 1.LFUK Motto : "The optimal health results from ensuring that the right
ové technologie v analýze D A, R A a proteinů Stanislav Kmoch Centrum aplikované genomiky, Ústav dědičných metabolických poruch, 1.LFUK Motto : "The optimal health results from ensuring that the right
Využití rekombinantní DNA při studiu mikroorganismů
 Využití rekombinantní DNA při studiu mikroorganismů doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2014 1 2 Obsah přednášky 1) Celogenomové metody sekvenování 2) Sekvenování H.
Využití rekombinantní DNA při studiu mikroorganismů doc. RNDr. Milan Bartoš, Ph.D. bartosm@vfu.cz Přírodovědecká fakulta MU, 2014 1 2 Obsah přednášky 1) Celogenomové metody sekvenování 2) Sekvenování H.
Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK
 MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
MODULARIZACE VÝUKY EVOLUČNÍ A EKOLOGICKÉ BIOLOGIE CZ.1.07/2.2.00/15.0204 Molecular Ecology J. Bryja, M. Macholán MU, P. Munclinger - UK Co je molekulární ekologie? Uměle vytvořený obor vymezený technickým
ZDRAVOTNÍ NEZÁVADNOST POTRAVIN
 ZDRAVOTNÍ NEZÁVADNOST POTRAVIN Možnosti stanovení Listeria monocytogenes popis metod a jejich princip Mária Strážiková Aleš Holfeld Obsah Charakteristika Listeria monocytogenes Listerióza Metody detekce
ZDRAVOTNÍ NEZÁVADNOST POTRAVIN Možnosti stanovení Listeria monocytogenes popis metod a jejich princip Mária Strážiková Aleš Holfeld Obsah Charakteristika Listeria monocytogenes Listerióza Metody detekce
4. Centrální dogma, rozluštění genetického kódu a zrod molekulární biologie.
 4. Centrální dogma, rozluštění genetického kódu a zrod molekulární biologie. Od genu k proteinu - centrální dogma biologie Geny jsou zakódovány v DNA - Jakým způsobem? - Jak se projevují? Již v roce 1902
4. Centrální dogma, rozluštění genetického kódu a zrod molekulární biologie. Od genu k proteinu - centrální dogma biologie Geny jsou zakódovány v DNA - Jakým způsobem? - Jak se projevují? Již v roce 1902
Pokročilé biofyzikální metody v experimentální biologii
 Pokročilé biofyzikální metody v experimentální biologii Ctirad Hofr 1/1 Proč biofyzikální metody? Biofyzikální metody využívají fyzikální principy ke studiu biologických systémů Poskytují kvantitativní
Pokročilé biofyzikální metody v experimentální biologii Ctirad Hofr 1/1 Proč biofyzikální metody? Biofyzikální metody využívají fyzikální principy ke studiu biologických systémů Poskytují kvantitativní
Sekvenování genomů. Human Genome Project: historie, výsledky a důsledky. MUDr. Jan Pláteník, PhD. Počátky sekvenování
 Sekvenování genomů Human Genome Project: historie, výsledky a důsledky MUDr. Jan Pláteník, PhD. (prosinec 2006) Počátky sekvenování 1965: přečtena sekvence trna kvasinky (80 bp) 1977: vynalezena Sangerova
Sekvenování genomů Human Genome Project: historie, výsledky a důsledky MUDr. Jan Pláteník, PhD. (prosinec 2006) Počátky sekvenování 1965: přečtena sekvence trna kvasinky (80 bp) 1977: vynalezena Sangerova
Buněčný cyklus. Replikace DNA a dělení buňky
 Buněčný cyklus Replikace DNA a dělení buňky 2 Regulace buněčného dělení buněčný cyklus: buněčné dělení buněčný růst kontrola kvality potomstva (dceřinných buněk) bránípřenosu nekompletně zreplikovaných
Buněčný cyklus Replikace DNA a dělení buňky 2 Regulace buněčného dělení buněčný cyklus: buněčné dělení buněčný růst kontrola kvality potomstva (dceřinných buněk) bránípřenosu nekompletně zreplikovaných
Zaměření bakalářské práce (témata BP)
 Zaměření bakalářské práce (témata BP) Obor: Buněčná a molekulární diagnostika - zadává katedra - studenti si témata losují Obor: molekulární biologie a genetika - témata BP vychází z vybraného tématu DP
Zaměření bakalářské práce (témata BP) Obor: Buněčná a molekulární diagnostika - zadává katedra - studenti si témata losují Obor: molekulární biologie a genetika - témata BP vychází z vybraného tématu DP
Návrh a implementace algoritmů pro adaptivní řízení průmyslových robotů
 Návrh a implementace algoritmů pro adaptivní řízení průmyslových robotů Design and implementation of algorithms for adaptive control of stationary robots Marcel Vytečka 1, Karel Zídek 2 Abstrakt Článek
Návrh a implementace algoritmů pro adaptivní řízení průmyslových robotů Design and implementation of algorithms for adaptive control of stationary robots Marcel Vytečka 1, Karel Zídek 2 Abstrakt Článek
Inovace studia molekulární a buněčné biologie
 Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
Investice do rozvoje vzdělávání Inovace studia molekulární a buněčné biologie Tento projekt je spolufinancován Evropským sociálním fondem a státním rozpočtem České republiky. Investice do rozvoje vzdělávání
CONTRIBUTION TO UNDERSTANDING OF CORRELATIVE ROLE OF COTYLEDON IN PEA (Pisum sativum L.)
 CONTRIBUTION TO UNDERSTANDING OF CORRELATIVE ROLE OF COTYLEDON IN PEA (Pisum sativum L.) PŘÍSPĚVEK K POZNÁNÍ KORLAČNÍ FUNKCE DĚLOHY U HRACHU (Pisum sativum L.) Mikušová Z., Hradilík J. Ústav Biologie rostlin,
CONTRIBUTION TO UNDERSTANDING OF CORRELATIVE ROLE OF COTYLEDON IN PEA (Pisum sativum L.) PŘÍSPĚVEK K POZNÁNÍ KORLAČNÍ FUNKCE DĚLOHY U HRACHU (Pisum sativum L.) Mikušová Z., Hradilík J. Ústav Biologie rostlin,
Rich Jorgensen a kolegové vložili gen produkující pigment do petunií (použili silný promotor)
 RNAi Rich Jorgensen a kolegové vložili gen produkující pigment do petunií (použili silný promotor) Místo silné pigmentace se objevily rostliny variegované a dokonce bílé Jorgensen pojmenoval tento fenomén
RNAi Rich Jorgensen a kolegové vložili gen produkující pigment do petunií (použili silný promotor) Místo silné pigmentace se objevily rostliny variegované a dokonce bílé Jorgensen pojmenoval tento fenomén
Laboratoř sekvenace DNA Servisní laboratoř biologické sekce PřF UK
 Laboratoř sekvenace DNA Založena 2003, momentálně jsme 2 zaměstnankyně, máme dvě spolupracovnice na DPP a jsme finančně soběstačné, včetně platů. Rok Počet sekvenací Počet fragmentací 2009 10 700 8 100
Laboratoř sekvenace DNA Založena 2003, momentálně jsme 2 zaměstnankyně, máme dvě spolupracovnice na DPP a jsme finančně soběstačné, včetně platů. Rok Počet sekvenací Počet fragmentací 2009 10 700 8 100
Genetické mapování. v přírodních populacích i v laboratoři
 Genetické mapování v přírodních populacích i v laboratoři Funkční genetika Cílem je propojit konkrétní mutace/geny s fenotypem Vzniklý v laboratoři pomocí mutageneze či vyskytující se v přírodě. Forward
Genetické mapování v přírodních populacích i v laboratoři Funkční genetika Cílem je propojit konkrétní mutace/geny s fenotypem Vzniklý v laboratoři pomocí mutageneze či vyskytující se v přírodě. Forward
V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014. Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU
 V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU Zemědělská 1, Budova A, 4. patro (učebny dle programu)
V. letní škola metod molekulární biologie nukleových kyselin a genomiky 16. - 20. 6. 2014 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU Zemědělská 1, Budova A, 4. patro (učebny dle programu)
Základy molekulární biologie KBC/MBIOZ
 Základy molekulární biologie KBC/MBIOZ 4. Metody molekulární biologie I Izolace DNA a RNA Specifické postupy pro baktérie, kvasinky, rostlinné a živočišné tkáně U RNA nutno zabránit kontaminaci RNasami
Základy molekulární biologie KBC/MBIOZ 4. Metody molekulární biologie I Izolace DNA a RNA Specifické postupy pro baktérie, kvasinky, rostlinné a živočišné tkáně U RNA nutno zabránit kontaminaci RNasami
Vyhledávání a charakteristika genů zodpovědných za modré zabarvení obilky pšenice seté (Triticum aestivum L.)
 Předběžná oponentura disertační práce Vyhledávání a charakteristika genů zodpovědných za modré zabarvení obilky pšenice seté (Triticum aestivum L.) Školitel: Prof. RNDr. Ladislav Havel, CSc. Doktorandka:
Předběžná oponentura disertační práce Vyhledávání a charakteristika genů zodpovědných za modré zabarvení obilky pšenice seté (Triticum aestivum L.) Školitel: Prof. RNDr. Ladislav Havel, CSc. Doktorandka:
Biotechnologický kurz. II. letní škola metod molekulární biologie nukleových kyselin a genomiky 17. - 21. 6. 2013
 Biotechnologický kurz Biotechnologický kurz II. letní škola metod molekulární biologie nukleových kyselin a genomiky 17. - 21. 6. 2013 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU v Brně Zemědělská
Biotechnologický kurz Biotechnologický kurz II. letní škola metod molekulární biologie nukleových kyselin a genomiky 17. - 21. 6. 2013 Ústav morfologie, fyziologie a genetiky zvířat AF MENDELU v Brně Zemědělská
Dědičnost pohlaví Genetické principy základních způsobů rozmnožování
 Dědičnost pohlaví Vznik pohlaví (pohlavnost), tj. komplexu znaků, vlastností a funkcí, které vymezují exteriérové i funkční diference mezi příslušníky téhož druhu, je výsledkem velmi komplikované série
Dědičnost pohlaví Vznik pohlaví (pohlavnost), tj. komplexu znaků, vlastností a funkcí, které vymezují exteriérové i funkční diference mezi příslušníky téhož druhu, je výsledkem velmi komplikované série
Analýza DNA. Co zjišťujeme u DNA DNA. PCR polymerase chain reaction. Princip PCR PRINCIP METODY PCR
 o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
o zjišťujeme u DN nalýza DN enetickou podstatu konkrétních proteinů Mutace bodové (sekvenční delece nebo inzerce nukleotidů), chromosomové aberace (numerické, strukturální) Polymorfismy konkrétní mutace,
2 Inkompatibilita v systému Rhesus. Upraveno z A.D.A.M.'s health encyclopedia
 2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
2 Inkompatibilita v systému Rhesus Upraveno z A.D.A.M.'s health encyclopedia 3 Inkompatibilita v systému Rhesus Úkol 7, str.119 Které z uvedených genotypových kombinací Rh systému u manželů s sebou nesou
DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU
 Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
Úvod IntellMed, s.r.o., Václavské náměstí 820/41, 110 00 Praha 1 DIAGNOSTICKÝ KIT PRO DETEKCI MINIMÁLNÍ REZIDUÁLNÍ CHOROBY U KOLOREKTÁLNÍHO KARCINOMU Jednou z nejvhodnějších metod pro detekci minimální
Mikrosatelity (STR, SSR, VNTR)
 Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
Mikrosatelity (STR, SSR, VNTR) Repeats Více než polovina našeho genomu Interspersed (transposony) Tandem (mini- a mikrosatelity) Minisatellites (longer motifs 10 100 nucleotides) mikrosatelity Tandemová
GENOTOXICITA A ZMĚNY V GENOVÉ EXPRESI
 GENOTOXICITA A ZMĚNY V GENOVÉ EXPRESI INDUKOVANÉ PŮSOBENÍM ORGANICKÝCH LÁTEK Z PRACHOVÝCH ČÁSTIC V OVZDUŠÍ Kateřina Hanzalová Oddělení genetické ekotoxikologie Ústav experimentální medicíny AV ČR v.v.i.
GENOTOXICITA A ZMĚNY V GENOVÉ EXPRESI INDUKOVANÉ PŮSOBENÍM ORGANICKÝCH LÁTEK Z PRACHOVÝCH ČÁSTIC V OVZDUŠÍ Kateřina Hanzalová Oddělení genetické ekotoxikologie Ústav experimentální medicíny AV ČR v.v.i.
Základy molekulární biologie KBC/MBIOZ
 Základy molekulární biologie KBC/MBIOZ Ivo Frébort 4. Metody molekulární biologie I Izolace DNA a RNA Specifické postupy pro baktérie, kvasinky, rostlinné a živočišné tkáně U RNA nutno zabránit kontaminaci
Základy molekulární biologie KBC/MBIOZ Ivo Frébort 4. Metody molekulární biologie I Izolace DNA a RNA Specifické postupy pro baktérie, kvasinky, rostlinné a živočišné tkáně U RNA nutno zabránit kontaminaci
NUKLEOVÉ KYSELINY. Základ života
 NUKLEOVÉ KYSELINY Základ života HISTORIE 1. H. Braconnot (30. léta 19. století) - Strassburg vinné kvasinky izolace matiére animale. 2. J.F. Meischer - experimenty z hnisem štěpení trypsinem odstředěním
NUKLEOVÉ KYSELINY Základ života HISTORIE 1. H. Braconnot (30. léta 19. století) - Strassburg vinné kvasinky izolace matiére animale. 2. J.F. Meischer - experimenty z hnisem štěpení trypsinem odstředěním
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ
 INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
INTRODUCING OF SNAPSHOT METHOD FOR POLYMORPHISM DETECTION ZAVEDENÍ SNAPSHOT METODIKY PRO DETEKCI POLYMORFISMŮ Civáňová K., Knoll A. Ústav genetiky, Agronomická fakulta, Mendelova zemědělská a lesnická
REKOMBINACE Přestavby DNA
 REKOMBINACE Přestavby DNA variace v kombinacích genů v genomu adaptace evoluce 1. Obecná rekombinace ( General recombination ) Genetická výměna mezi jakýmkoli párem homologních DNA sekvencí - často lokalizovaných
REKOMBINACE Přestavby DNA variace v kombinacích genů v genomu adaptace evoluce 1. Obecná rekombinace ( General recombination ) Genetická výměna mezi jakýmkoli párem homologních DNA sekvencí - často lokalizovaných
2. Z následujících tvrzení, týkajících se prokaryotické buňky, vyberte správné:
 Výběrové otázky: 1. Součástí všech prokaryotických buněk je: a) DNA, plazmidy b) plazmidy, mitochondrie c) plazmidy, ribozomy d) mitochondrie, endoplazmatické retikulum 2. Z následujících tvrzení, týkajících
Výběrové otázky: 1. Součástí všech prokaryotických buněk je: a) DNA, plazmidy b) plazmidy, mitochondrie c) plazmidy, ribozomy d) mitochondrie, endoplazmatické retikulum 2. Z následujících tvrzení, týkajících
RESEARCH OF ANAEROBIC FERMENTATION OF ORGANIC MATERIALS IN SMALL VOLUME BIOREACTORS
 RESEARCH OF ANAEROBIC FERMENTATION OF ORGANIC MATERIALS IN SMALL VOLUME BIOREACTORS Trávníček P., Vítěz T., Dundálková P., Karafiát Z. Department of Agriculture, Food and Environmental Engineering, Faculty
RESEARCH OF ANAEROBIC FERMENTATION OF ORGANIC MATERIALS IN SMALL VOLUME BIOREACTORS Trávníček P., Vítěz T., Dundálková P., Karafiát Z. Department of Agriculture, Food and Environmental Engineering, Faculty
Co zjišťujeme u DNA ACGGTCGACTGCGATGAACTCCC ACGGTCGACTGCGATCAACTCCC ACGGTCGACTGCGATTTGAACTCCC
 Analýza DNA Co zjišťujeme u DNA genetickou podstatu konkrétních proteinů mutace bodové, sekvenční delece/inzerce nukleotidů, chromosomové aberace (numerické, strukturální) polymorfismy konkrétní mutace,
Analýza DNA Co zjišťujeme u DNA genetickou podstatu konkrétních proteinů mutace bodové, sekvenční delece/inzerce nukleotidů, chromosomové aberace (numerické, strukturální) polymorfismy konkrétní mutace,
RIGORÓZNÍ OTÁZKY - BIOLOGIE ČLOVĚKA
 RIGORÓZNÍ OTÁZKY - BIOLOGIE ČLOVĚKA 1. Genotyp a jeho variabilita, mutace a rekombinace Specifická imunitní odpověď Prevence a časná diagnostika vrozených vad 2. Genotyp a prostředí Regulace buněčného
RIGORÓZNÍ OTÁZKY - BIOLOGIE ČLOVĚKA 1. Genotyp a jeho variabilita, mutace a rekombinace Specifická imunitní odpověď Prevence a časná diagnostika vrozených vad 2. Genotyp a prostředí Regulace buněčného
Osekvenované genomy. Pan troglodydes, 2005. Neandrtálec, 2010
 GENOMOVÉ PROJEKTY Osekvenované genomy Haemophilus influenze, 1995 první osekvenovaná bakterie Saccharomyces cerevisiae, 1996 první osekvenovaný eukaryotický organimus Caenorhabditis elegans, 1998 první
GENOMOVÉ PROJEKTY Osekvenované genomy Haemophilus influenze, 1995 první osekvenovaná bakterie Saccharomyces cerevisiae, 1996 první osekvenovaný eukaryotický organimus Caenorhabditis elegans, 1998 první
Příprava vektoru IZOLACE PLASMIDU ALKALICKÁ LYZE, KOLONKOVÁ IZOLACE DNA GELOVÁ ELEKTROFORÉZA RESTRIKČNÍ ŠTĚPENÍ. E. coli. lyze buňky.
 Příprava vektoru IZOLCE PLSMIDU LKLICKÁ LYZE, KOLONKOVÁ IZOLCE DN E. coli plasmidová DN proteiny proteiny + + vysrážená plasmidová lyze buňky + snížení ph chromosomální DN centrifugace DN chromosomální
Příprava vektoru IZOLCE PLSMIDU LKLICKÁ LYZE, KOLONKOVÁ IZOLCE DN E. coli plasmidová DN proteiny proteiny + + vysrážená plasmidová lyze buňky + snížení ph chromosomální DN centrifugace DN chromosomální
